近期再一次回顾和学习了下 SNP 芯片 HLA 填充,发现近两年有几个新的软件出来,文章发表的也不错,在这里分享一下!
CookHLA[1]
与以前的方法相比,CookHLA 大大提高了插补的准确性,包括通过实施多项更改的前身 SNP2HLA。首先,为了提高准确性和效率,我们采用了最近发布的隐马尔可夫模型(Beagle v4 和 v5)。其次,为了进一步提高准确性,我们开发了一种可以解释 HLA 基因外显子局部变异的程序。高度多态性外显子(I 类中的外显子 2/3/4 和 II 类中的 2/3)是关键区域,其序列可以决定大多数 4 位等位基因。在我们的方法中,我们通过将标记集局部放置在每个高度多态的外显子中来重复插补,并使用重复分析的共识后验概率进行最终预测(图 B)。第三,为了进一步提高准确性,我们从数据中自适应地学习 MHC 的遗传图谱。该图谱信息使我们能够解释 MHC 内特定于数据的 LD 结构,与使用公开的遗传图谱相比,这提高了插补的准确性(图 C)。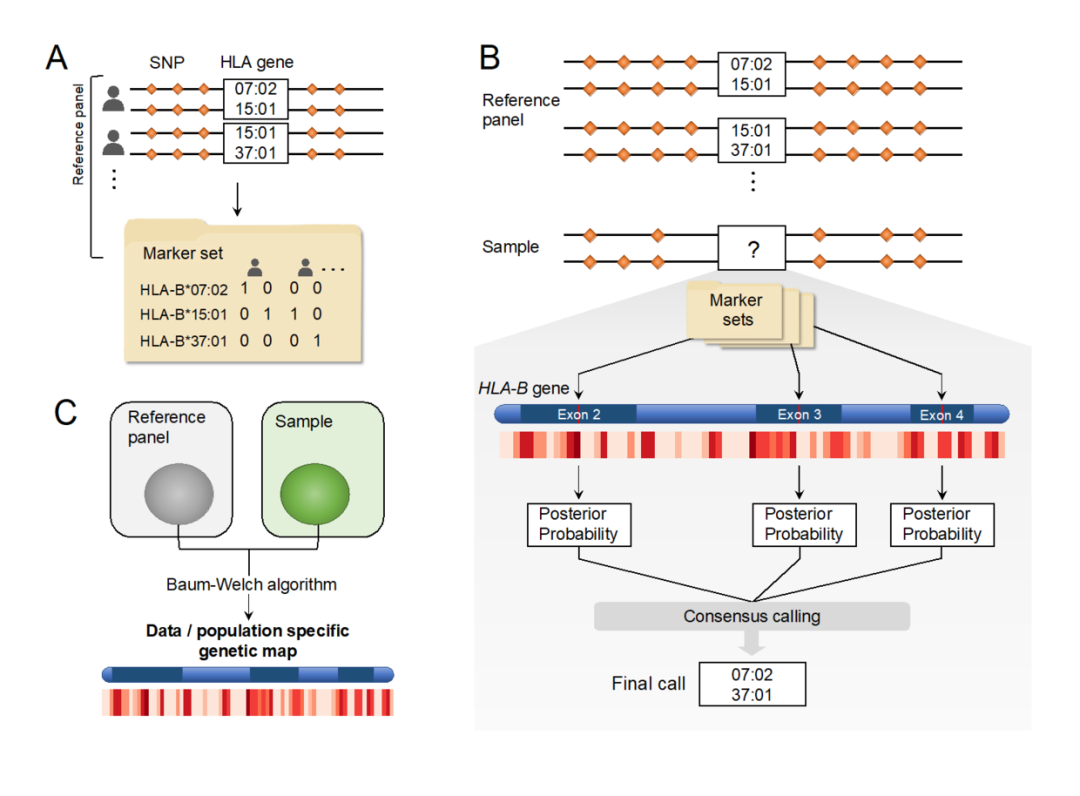 个人测试发现它的缺点是多了一步,增加了参考生成的难度和出错的可能性。
个人测试发现它的缺点是多了一步,增加了参考生成的难度和出错的可能性。
DEEP-HLA[2]
 DEEP*HLA 是一种基于 Python 中实现的多任务卷积神经网络的 HLA 等位基因插补方法。
DEEP*HLA 是一种基于 Python 中实现的多任务卷积神经网络的 HLA 等位基因插补方法。
DEEP*HLA 接收预分期 SNV 数据并输出二元 HLA 等位基因的基因型剂量。
在 DEEP*HLA 中,HLA 插补在两个过程中形成:
(1) 使用 HLA 参考面板进行模型训练
(2)使用经过训练的模型进行插补。最近大火的 chatGPT 让每个人了解了 AI 正在成为我们的好(取)帮(代)手(者),有人说打败我们的不是 AI,是使用 AI 的人,所以用起这个 AI 的 HLA 工具吧!个人体验,软件安装依赖有问题,这也是开源软件的弊端啦!
HLA-TAPAS[3]
 HLA-TAPAS(HLA-Type at Protein for Association Studies)是一个以 HLA 为中心的管道,可以处理 HLA 参考面板构建(MakeReference),HLA 插补(SNP2HLA)和 HLA 关联(HLAassoc)。
HLA-TAPAS(HLA-Type at Protein for Association Studies)是一个以 HLA 为中心的管道,可以处理 HLA 参考面板构建(MakeReference),HLA 插补(SNP2HLA)和 HLA 关联(HLAassoc)。
简而言之,主要更新包括
(1) 使用 PLINK-v1.9 代替 v1.07;
(2) 使用 BEAGLE v4.1 代替 v3 进行定相和插补;和
(3) 包括自定义 R 脚本,用于在多个祖先中执行关联和精细映射分析。
个人体会这个软件是一个流程,比较全面的解决方案,还未试用!
Michigan Imputation Server[4]
多种族 HLA 小组由五个全球种群的 36,586 个单倍型组成。我们以 two-field (four-digit) and G-group 分辨率发布了该面板。
个人感觉这个的缺点是多数是欧美人,中国人的数据比较少。
PGG.MHC–人类主要组织相容性复合体基因数据库和分析平台
这个前面提过,这里不再重复!PGG.MHC–人类主要组织相容性复合体基因数据库和分析平台
参考资料
CookHLA: https://github.com/WansonChoi/CookHLA
[2]DEEP-HLA: https://github.com/tatsuhikonaito/DEEP-HLA
[3]HLA-TAPAS: https://github.com/immunogenomics/HLA-TAPAS
[4]Michigan Imputation Server: https://imputationserver.sph.umich.edu/index.html#!pages/home
[5]PGG.MHC–人类主要组织相容性复合体基因数据库和分析平台: https://www.jianshu.com/p/dc93acb0110e
本篇文章来源于微信公众号:微因