划重点:
-
最新版本的Greengenes2数据库 -
新的可视化工具 da_barplot
计划的下一个QIIME 2版本计划于2023年5月发布(QIIME2 2023 5),但请继续关注更新。
查看QIIME2 2023.2 文档[1]有关安装最新 QIIME2 版本的详细信息,以及教程和其他资源。如果您遇到任何问题,请在QIIME 2论坛上与QIIME2官方团队联系!
Docker 镜像已经构建完毕,可以使用了!
以下是该版本的亮点:
-
q2-composition[2]
-
每个选项卡现在都包含 LFC 和 W-scores的数据切片名称和简短描述
-
用于计算截距的列包含在每个切片表的顶部
-
修复了 ANCOM-BC
reference_level中的一个bug,其中选择的值是元数据中的列,但在特征表中 找不到该组中的关联 ID 不会引发任何错误,ANCOM-BC 只是默认恢复截距列的字母顺序。现在,这将产生一个reference_level包含该值的错误, 以及表中未找到的ID -
为参数添加了类型强制,现在只允许使用数字元数据列
reference_level -
增加了对ANCOM和ANCOM-BC方法的引用
-
对
tabulate可视化工具进行了一些改进: -
添加了一个名为**
da_barplot的新可视化工具””,该工具添加了对从 ANCOM-BC 数据生成条形图的支持,灵感来自图6[3]在ANCOM-BC论文中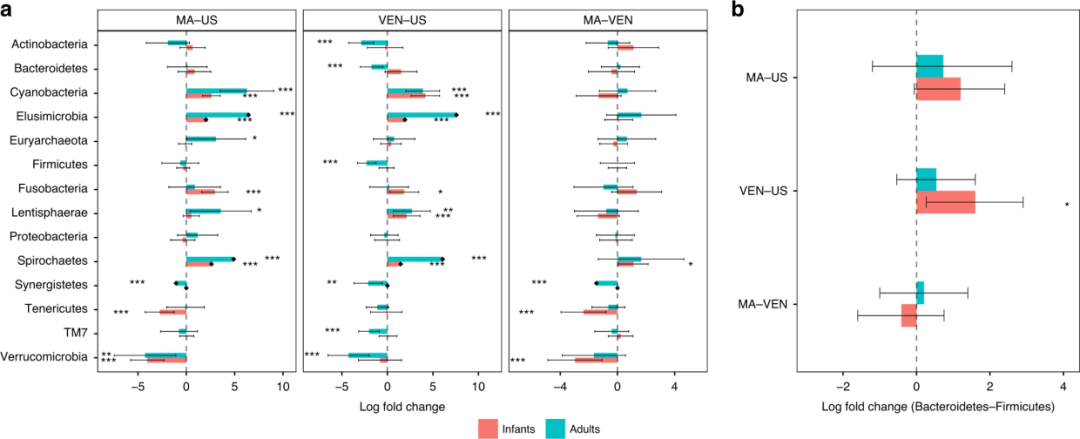
-
在 ANCOM 中添加了一个名为
--filter-missing的新标志,该标志将在启用时过滤缺少元数据的示例,如果禁用,将产生一个错误,其中包含缺少元数据的ID -
q2-feature-table[4]
-
编写了一个更改,使用于** summarize输出的语言更加清晰**,将特征计数更改为频率 -
q2-fmt[5]
-
增加了一种供体菌株比例植入(PEDS)方法 sample_peds,称为跟踪FMT干预后转移到受体的供体特征的百分比该方法在粪便微生物群移植后细菌菌株的精确定量描绘了长期植入并解释了结果 |自然微生物学[6]. -
添加了一个名为 plot_heatmap的新可视化工具,该可视化工具增加了对生成热图的支持,以随着时间的推移可视化每个主题的PEDS -
q2-stats[7]
-
修复了由 frictionless依赖项的 API 更改导致的一些相关测试失败bugs -
q2-types[8]
-
在更新 per_sample_sequences调用的格式中进行了小更新(从.iteritems到.items),因为pandas 将在不久的将来弃用.iteritems -
文档[9]
-
修改了Greengenes条目,现在包括 2022.10,即最新版本的Greengenes2[10]
参考资料
QIIME2 2023.2 文档: https://docs.qiime2.org/2023.2/
[2]q2-composition: https://github.com/qiime2/q2-composition
[3]图6: https://www.nature.com/articles/s41467-020-17041-7/figures/6
[4]q2-feature-table: https://github.com/qiime2/q2-feature-table
[5]q2-fmt: https://github.com/qiime2/q2-fmt
[6]粪便微生物群移植后细菌菌株的精确定量描绘了长期植入并解释了结果 |自然微生物学: https://www.nature.com/articles/s41564-021-00966-0
[7]q2-stats: https://github.com/qiime2/q2-stats
[8]q2-types: https://github.com/qiime2/q2-types
[9]文档: https://github.com/qiime2/docs
[10]最新版本的Greengenes2: https://forum.qiime2.org/t/introducing-greengenes2-2022-10/25291
本篇文章来源于微信公众号:微因