我们知道,宏基因组中krona图来展示某个样品的详细信息,最近有点好奇16S的进化树可视化问题,这不,在qiime2官方视频教程里发现了一个方法,很不错,分享下!官方视频教程我搬到B站啦,在AI的加持下加了字幕,并精校了错误。18 Phylogenetic tree construction[1]
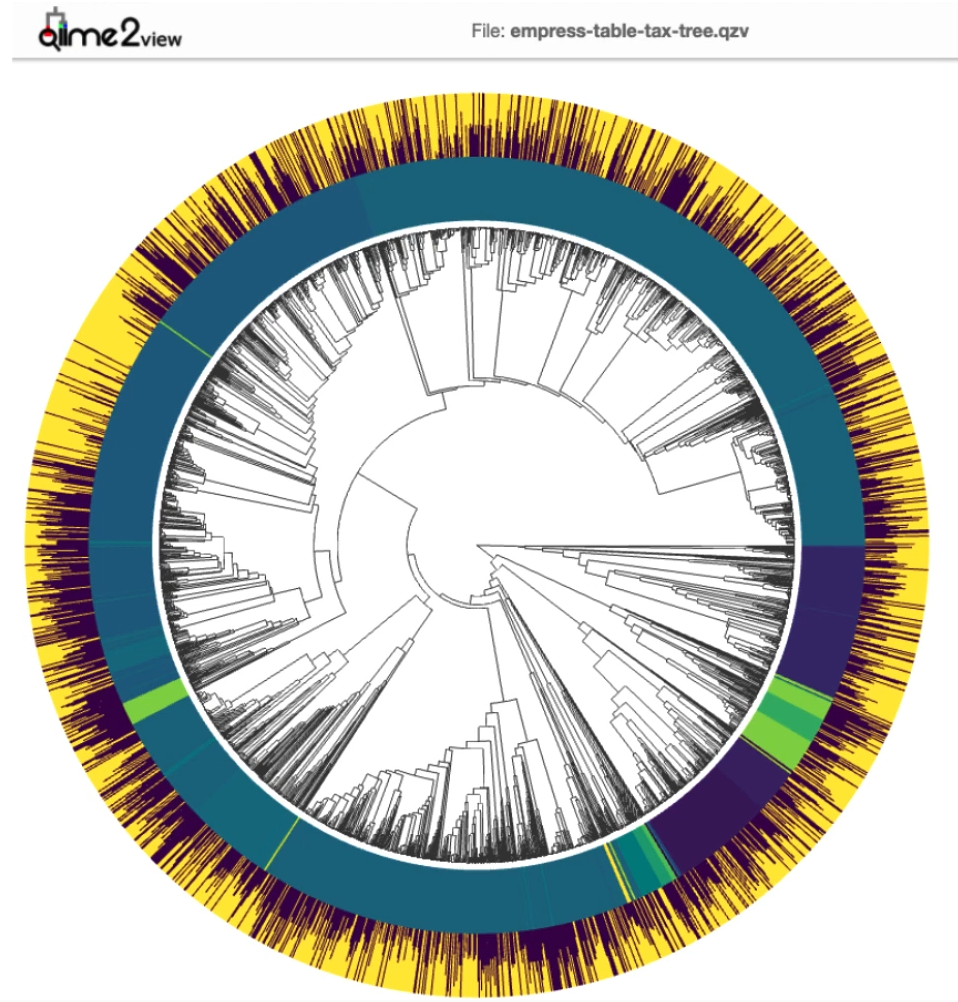
先欣赏下图的效果啦,还不错吧,进化树+丰度的展示,有点circos那个味了呢。下面,就看下怎样做出这个图(以下来自官方教程biocore/empress[2]):
介绍
一个快速且可扩展的系统发育树[3]查看器。Empress帮助用户探索数据集中特征之间的层次结构关系。任何类型的“特征”都可以通过这种方式查看:从历史上看,这些特征通常代表群落调查中物种的进化关系,但您可以通过分层组织查看几乎任何类型的信息。例如,我们可以查看从 16S rRNA 标记基因测序数据生成的扩增子序列变异 (ASV)[4] 或操作分类单元 (OTU)[5] 树、从鸟枪法宏基因组学测序数据生成的树或使用 Qemistree[6] 生成的代谢组学数据树(仅举几个选项)。
Empress支持绝对新的功能,例如与命令图的集成和同步动画,以及已建立的树查看器通用的功能(例如元数据着色,分支折叠和条形图)。
截图
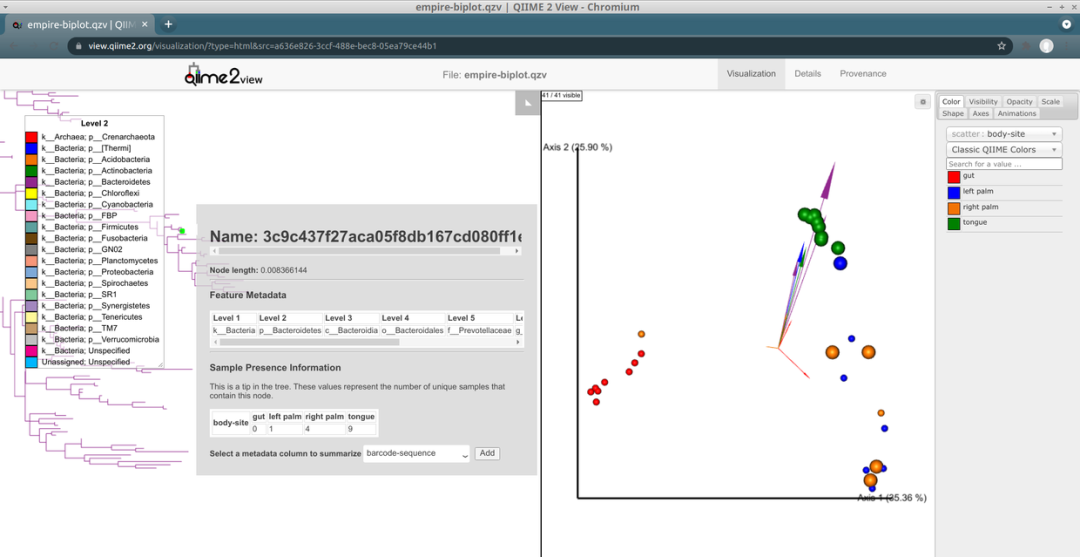
“empire”可视化了Empress扩增子序列变异(ASV)的系统发育树(左),以及empire的PCoA双标图(右)。作为集成这些显示器的方式之一,在树中选择一个尖端(代表 ASV)会放大 Emperor 中含有该 ASV 的样本——从而提供比单独使用任何显示器提供的信息更多的信息。
安装和基本用法
Empress可以作为独立程序或QIIME 2插件使用。独立版本将生成一个文件夹,其中包含查看绘图所需的 HTML/JS/CSS 文件,而 QIIME 2 版本将生成一个可视化,可以在 https://view.qiime2.org/[7] 或使用 ..qzv``qiime tools view
如果您在尝试安装 Empress 时遇到问题,请查看文档中的“安装疑难解答”部分[8]。(如果这些信息还不够,请随时在此存储库中提出问题[9]或在QIIME 2论坛[10]上发布问题!
独立版本
Empress可通过PyPI[11]获得。我们建议使用至少 3.6 的 Python 版本将 Empress 安装到环境(例如 conda[12] 环境)中。
运行以下命令以安装empress:
pip install cython "numpy >= 1.12.0"
pip install empress
尝试运行empress --help命令以确保已正确安装Empress。如果您看到不同Empress命令的详细信息,则安装已成功,您已准备好开始使用Empress!
可用命令
Empress提供两个命令:empress tree-plot和empress community-plot 。这两个命令都会生成 Empress 可视化,但community-plot需要您传入特征表和示例元数据,而tree-plot只需要一个树文件。有关更多详细信息,请参阅文档的此部分[13]。
输入文件
Empress的独立版本采用以下文件类型作为输入。(请注意,除树之外的所有这些都是可选的,对于empress tree-plot除树之外的所有内容是可选的,对于empress community-plot是除了树,特征表和示例元数据都是可选的。
| 输入 | 文件类型 |
|---|---|
| 树 | Newick[14] |
| Feature Table | BIOM[15] |
| Sample Metadata | TSV[16] |
| Feature Metadata | TSV[17] |
| PCoA | scikit-bio orddinationResults[18] |
独立用法示例
empress tree-plot
# Option 1: Using "long" parameter names
empress tree-plot
--tree tree.nwk
--feature-metadata feature-metadata.tsv
--output-dir tree-viz
# Option 2: Using "short" parameter names
empress tree-plot -t tree.nwk -fm feature-metadata.tsv -o tree-viz
empress community-plot
# Option 1: Using "long" parameter names
empress community-plot
--tree tree.nwk
--table feature-table.biom
--sample-metadata sample-metadata.tsv
--feature-metadata feature-metadata.tsv
--pcoa ordination.txt
--filter-extra-samples
--output-dir community-tree-viz
# Option 2: Using "short" parameter names
empress community-plot
-t tree.nwk
-tbl feature-table.biom
-sm sample-metadata.tsv
-fm feature-metadata.tsv
-p ordination.txt
--filter-extra-samples
-o community-tree-viz
您可以使用empress tree-plot --help和empress community-plot --help查看命令行参数的详细信息。请注意,提供给 -o/--output-dir 的路径不得存在,因为它将在成功执行命令后由 Empress 创建。还值得注意的是,Empress 命令的独立版本不支持提供多个样本/特征元数据文件。例如,如果您有多个要素元数据文件,则应将它们全部合并到一个传递给 Empress 的文件中。
输出将是一个包含empress.html文件的目录和一个包含在浏览器中查看绘图所需的 JS/CSS 文件的support_files目录。如果您为community-plot命令提供了 PCoA,则还将有一个emperor-resources子目录,其中包含在树旁边查看皇帝情节所需的文件。您可以在任何现代浏览器中查看empress.html文件,以与QIIME 2可视化相同的方式与之交互。
Qiime2版
有关如何安装 QIIME 2 的说明,请参阅 QIIME 2 安装[19]页面。安装 QIIME 2 后,请确保通过运行以下命令激活 conda 环境:
conda activate qiime2-2020.8
您可以将上述内容替换为您当前安装的任何版本的QIIME 2。qiime2-2020.8
现在,运行以下命令以使用 PyPI 安装 Emqueen:
pip install empress
安装empress,运行以下命令以确保empress安装正确。如果您看到有关Empress的QIIME 2插件的信息,则安装成功!
qiime dev refresh-cache
qiime empress --help
QIIME 2 用法示例
qiime empress tree-plot
qiime empress tree-plot
--i-tree tree.qza
--m-feature-metadata-file taxonomy.qza
--o-visualization tree-viz.qzv
qiime empress community-plot
qiime empress community-plot
--i-tree tree.qza
--i-feature-table feature-table.qza
--m-sample-metadata-file sample-metadata.tsv
--m-feature-metadata-file taxonomy.qza
--i-pcoa ordination.qza
--p-filter-extra-samples
--o-visualization community-tree-viz.qzv
参考资料
18 Phylogenetic tree construction: https://www.bilibili.com/video/BV1Re4y1B7ku/?share_source=copy_web&vd_source=fb425d6f0ad40ac954b4bdf7090a013f
[2]
biocore/empress: https://github.com/biocore/empress
[3]
的系统发育树: https://en.wikipedia.org/wiki/Phylogenetic_tree
[4]
扩增子序列变异 (ASV): https://en.wikipedia.org/wiki/Amplicon_sequence_variant
[5]
操作分类单元 (OTU): https://en.wikipedia.org/wiki/Operational_taxonomic_unit
[6]
Qemistree: https://github.com/biocore/q2-qemistree
[7]
https://view.qiime2.org/: https://view.qiime2.org/
[8]
“安装疑难解答”部分: https://github.com/biocore/empress#installation-troubleshooting
[9]
提出问题: https://github.com/biocore/empress/issues
[10]
QIIME 2论坛: https://forum.qiime2.org/
[11]
PyPI: https://pypi.org/project/empress/
[12]
conda: https://docs.conda.io/
[13]
此部分: https://github.com/biocore/empress#first-a-note-about-empress-commands
[14]
Newick: https://en.wikipedia.org/wiki/Newick_format
[15]
BIOM: http://biom-format.org/
[16]
TSV: https://en.wikipedia.org/wiki/Tab-separated_values
[17]
TSV: https://en.wikipedia.org/wiki/Tab-separated_values
[18]
scikit-bio orddinationResults: http://scikit-bio.org/docs/latest/generated/skbio.io.format.ordination.html
[19]
QIIME 2 安装: https://docs.qiime2.org/2020.8/install/
本篇文章来源于微信公众号: 微因