最近这波疫情,重现当年初中非典时期,甚至愈演愈烈,与之前初中时的封校住宿学习不同,已经工作的今天和太多的互联网信息大爆炸让我们有些焦虑,特别是,作为学习生物的人,我们也感到无能为力。官方媒体的科普,已经让大家对这个病毒的具体情况有所了解。我注意到,NJEM也已经把许多文章翻译成了中文版,以正视听。在这个时候,我们不能听信谣言!那么作为有些生物学素养的我们,也应该以自己的知识,学习下这个病毒的信息,以我们自己的理解!
下面是我的小小探索,虽然不足挂齿,至少我开启了探索之路。想起了那句名言,只要开始,便会心生热忱,持续为之,便能走向成功!(来自一本影响了我一生的书《方与圆》–丁远峙)
在这里放上几篇NJEM文章的中文翻译:
>>NEJM官方翻译 | 美国首例新冠病毒肺炎的治疗过程
>>重要数据|中国团队《新英格兰医学杂志》发布新型冠状病毒研究
>>新型冠状病毒上海救治专家组组长张文宏:病毒解码后的科学防控
>>中国国际电视台专访NEJM主编、传染病专家:严阵以待新型肺炎
1.序列上的一些探索
参考自https://github.com/BenLangmead/ads1-notebooks
def readGenome(filename):
#读取全部序列
genome = ''
with open(filename, 'r') as f:
for line in f:
# ignore header line with genome information
if not line[0] == '>':
genome += line.rstrip()
return genome
genome = readGenome('../Wuhan-RNA.fasta')
import collections
collections.Counter(genome)
#Counter({'A': 8954, 'U': 9594, 'G': 5863, 'C': 5492})
import matplotlib.pyplot as plt
def findGCByPos(file):
''' Find the GC ratio at each position in the read '''
# Keep track of the number of G/C bases and the total number of bases at each position
gc = [0] * 70
totals = [0] * 71
with open(file) as f:
for line in f:
#print(len(line))
if line.startswith('>'):
continue
for i in range(0,len(line)):
if line[i] == 'C' or line[i] == 'G':
gc[i] += 1
totals[i] += 1
#print(len(line))
# Divide G/C counts by total counts to get the average at each position
for i in range(len(gc)):
if totals[i] > 0:
gc[i] /= float(totals[i])
#print(totals)
return gc
gc = findGCByPos('../Wuhan-RNA.fasta')
plt.plot(range(len(gc)), gc)
plt.show()
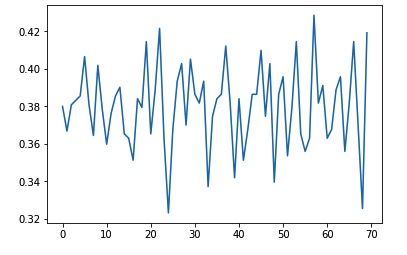
这些代码后,就可以看到GC含量,是以每行的每个碱基集合为一组来比较的,应该也比较有说服力,只有40左右。
2.RNA折叠
又想到怎么探索下这一串序列在病毒壳里面是怎么折叠的呢?找到了两个软件,首先是Vienna RNAfold,自己运行了一下,花费一两个小时,好像结果不咋地。
#安装这个软件,还是conda
conda install -c bioconda viennarna
RNAfold < Wuhan-RNA.fasta > Wuhan-rna.ss
RNAplot > Wuhan-rna.ss
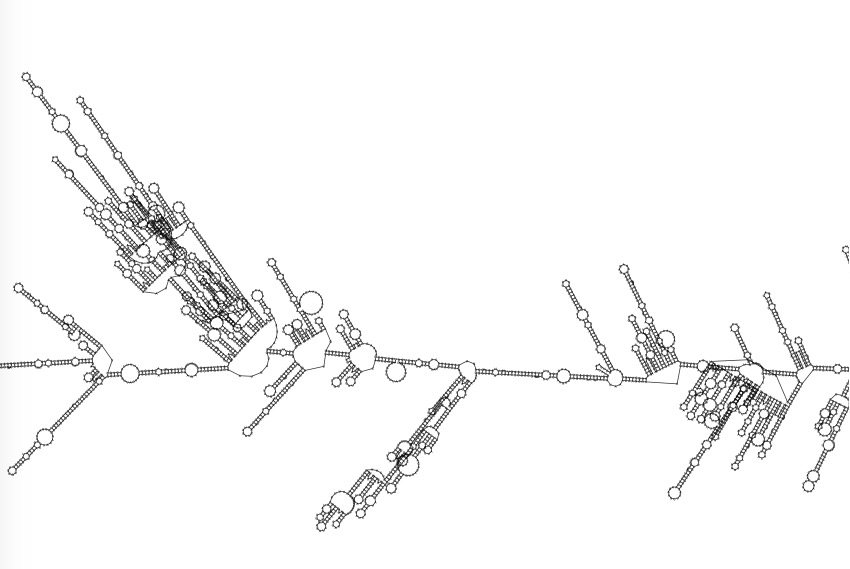
乍一看像一个进化树,如图:

放大了看,是颈环结构,发展一些地方折叠的并不好。
3.百度linearFold网页服务试用
偶然搜索发现百度也开发了个加速算法,速度提升很多,计算时间从小时级别到秒级别,测试确实如此,一分钟内返回了结果,看了下:

连线应该表示的是连接, forna (Kerpedjiev et al 2015) 可视化,由于长度太长,没法画出,后面尝试用本地画试试。