1.Feature表统计
继续我的qiime2图形界面q2studio学习笔记,之前已经获得了featuretable,现在对这个表做个统计。依旧是很简单的,选择featuretable–visualization-summarize table,这里是需要实验设计文件(metadata)的,最好放上,这里我就不放了。点run之后,很快就有结果了。
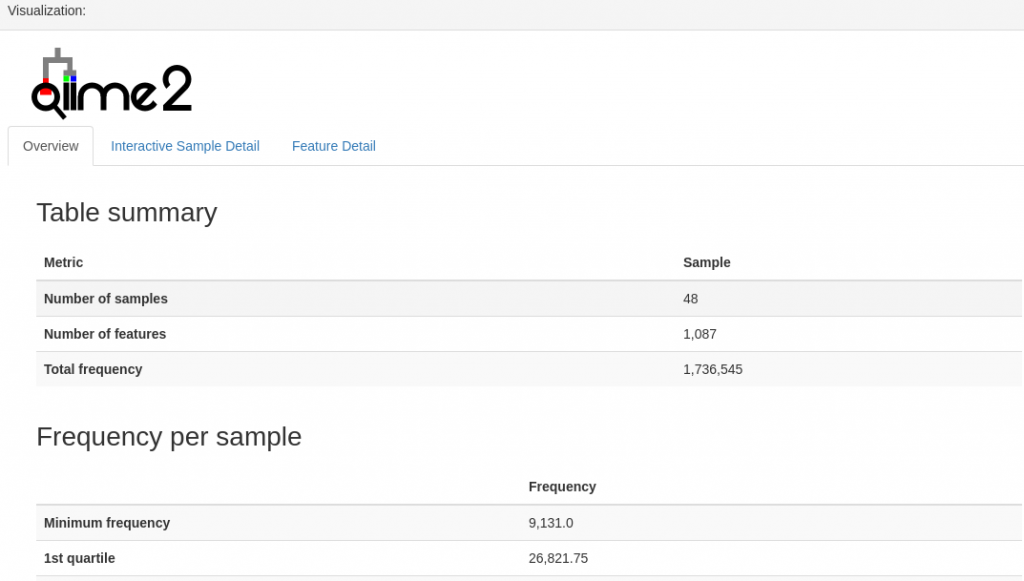
结果就像教程里的那样,
2.代表序列统计
这也没什么好说的,统计一下代表性的序列。使用feature taable里的view sequence associated with each feature选项,然后程序会自动载入数据,执行即可。
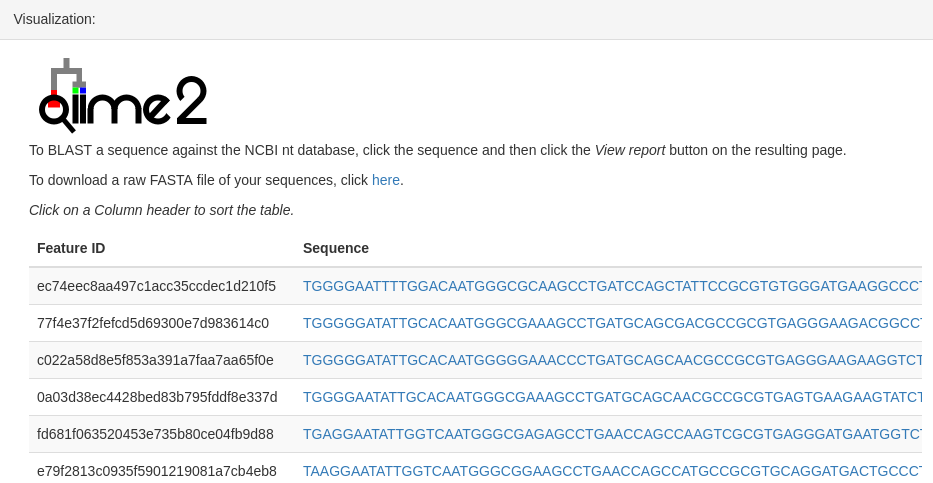
结果如图:
3.建树:用于多样性分析
3.1 多序列比对
alignment–de nova multiple alignment with mafft, 中间不小心报错,原因不明,但显示已完成,先放这。
3.2 移除高变区
应该是这个alignment–Positional conservation and gap filtering.默认参数试试,因为命令行没有给出参数。
3.3 建树
phylogeny–construct a phylogenetic tree with FastTree
3.4 无根树转换为有根树
phylogeny–midpoint root an rooted phylogenetic tree
4.计算多样性(包括所有常用的Alpha和Beta多样性方法),输入有根树、Feature表、样本重采样深度(一般为最小样本数据量,或覆盖绝大多数样品的数据量)
diversity–Core Diversity metrics(phylogeny and non phylogeny)
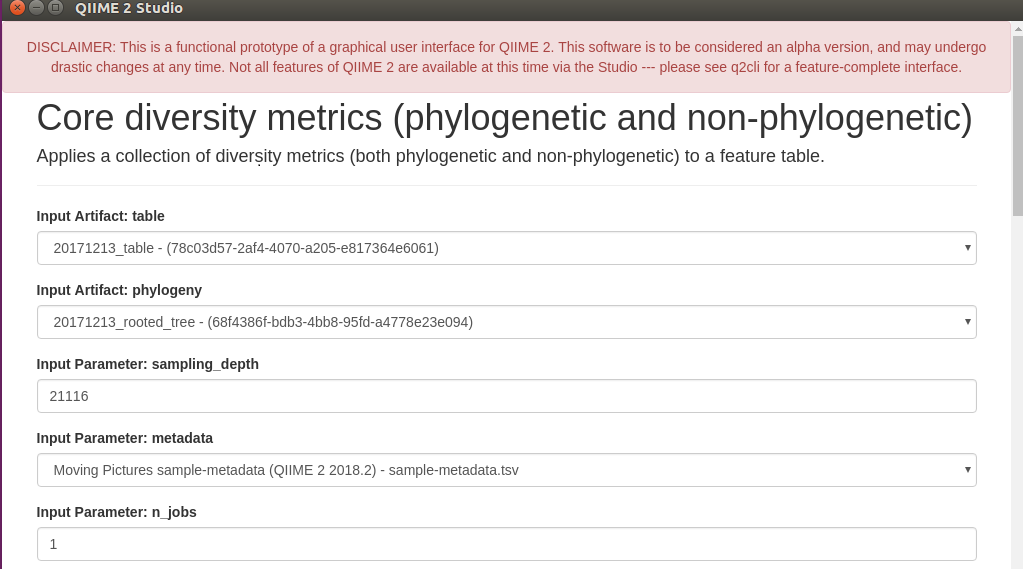
因为手头的数据不清楚实验分组情况,随便做了个metadata,只包含样本名称和分组信息的文件。
果然出现了报错:
/home/qiime2/miniconda/envs/qiime2-2018.4/lib/python3.5/site-packages/sklearn/utils/validation.py:475: DataConversionWarning: Data with input dtype int64 was converted to bool by check_pairwise_arrays. warnings.warn(msg, DataConversionWarning)
5.物种分类
5.1 物种注释
鉴于出现了错误,估计beta多样性也做不起来,那么就先看下特种注释,这个对我来说目前最有价值的数据。
下载注释所需文件:
https://data.qiime2.org/2018.4/common/gg-13-8-99-515-806-nb-classifier.qza
下载了个旧版本2017。6的文件报错不兼容,软件兼容性啊,也是醉了,于是下这个2018。4。
feature-classifier–classify-sklearn
5.2 物种分类柱状图
taxa–visualization–Visualize taxonomy with an interactive bar plot 是交互式的动态图哦!
当当当当,让人魂牵梦莹的图终于出现了,今天的学习先到这里。
