
摘要
噬菌体在微生物群落中多种多样且丰富,它们在微生物群落的进化和适应中发挥着重要作用。噬菌体复制和繁殖通常被认为限制在单个或狭窄的宿主范围内。法国巴斯德研究所的研究人员,使用来自各种环境的已发表和新生成的基于邻近连接的宏基因组 Hi-C(metaHiC)数据来探索病毒与宿主的相互作用。重建了 4,975 个中等质量或更高的微生物基因组和 6,572 个噬菌体基因组。MetaHiC 在基因组之间产生了联系网络,并能够将大约一半的噬菌体基因组分配给其宿主,结果表明这些噬菌体中有很大一部分在海洋或人类肠道等不同环境中与多个物种相互作用。这一观察结果挑战了噬菌体宿主范围狭窄的传统观点,揭示了多宿主关联在生态系统中很常见,并对它们可能如何影响生态学和进化以及噬菌体治疗方法产生影响。
主要结果
来自宏基因组邻近连接数据的病毒分箱
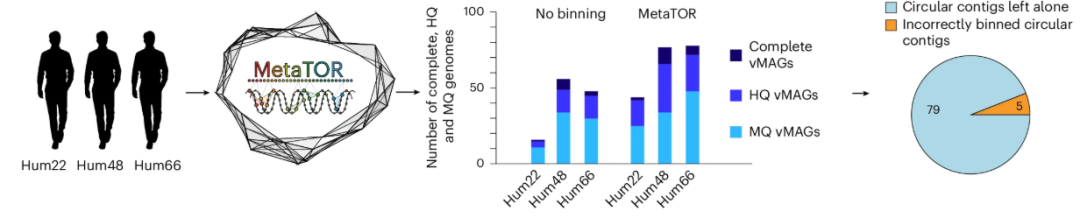
结果表明,该方法对病毒重叠群进行了适当的分箱,与SemBin或ViralCC 产生的高度污染的 vMAG 形成鲜明对比。MetaTOR 从单个宏基因组样本中生成了数十个具有完整单个病毒基因组的所有特征的 vMAG。它还强调了对接触图进行目视检查以正确评估邻近连接数据集的基因组重建质量的价值。而且与原始组装和单个重叠群的评估相比,MetaTOR显着增加了检索到的完整、HQ和MQ病毒基因组的数量。
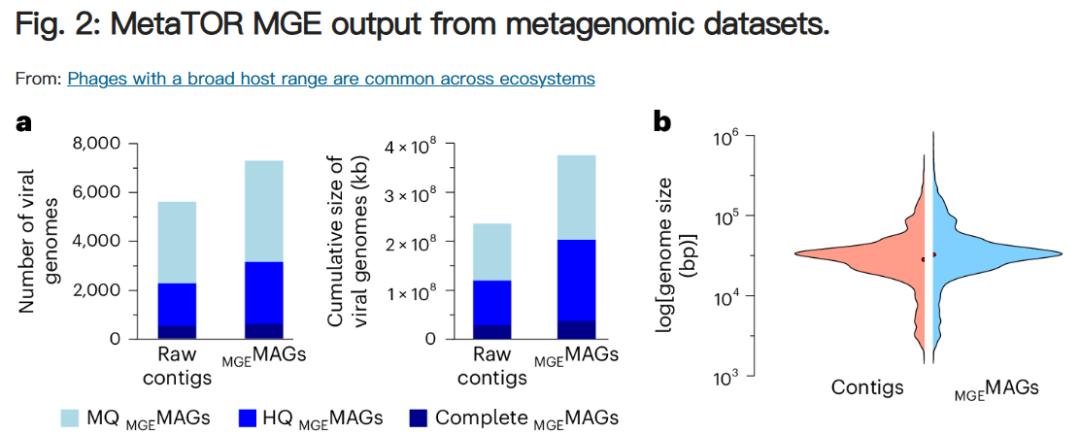
很大一部分病毒表现出多个宿主
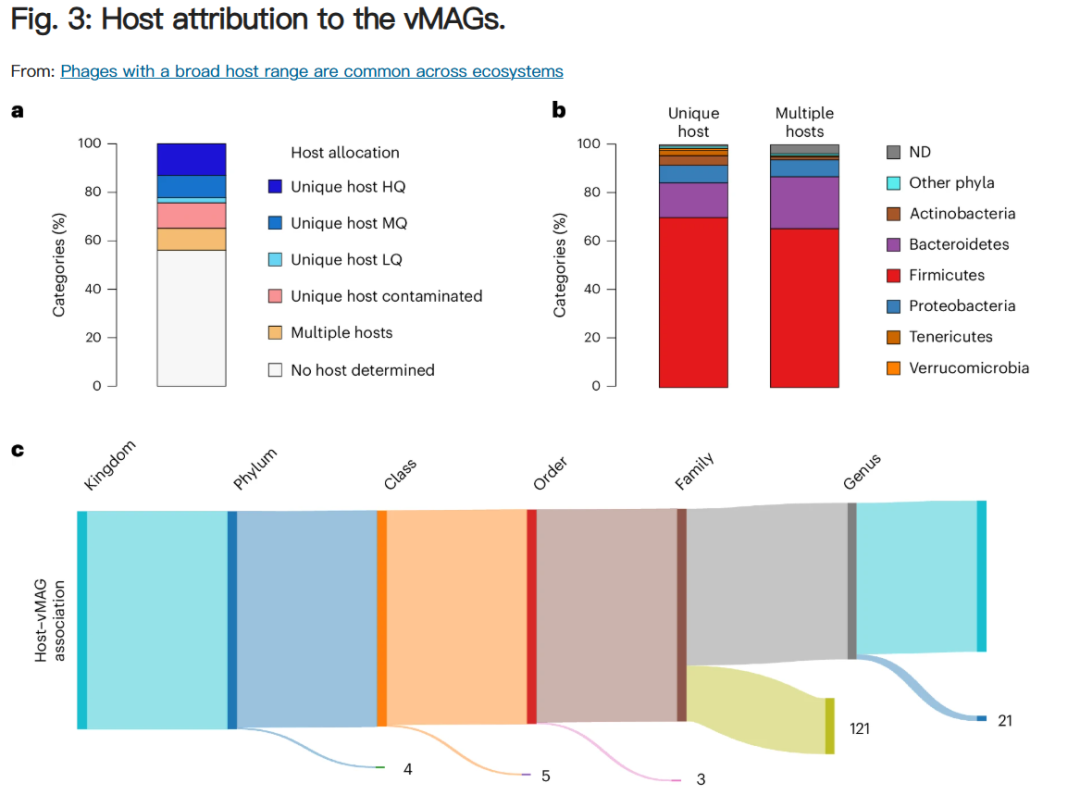
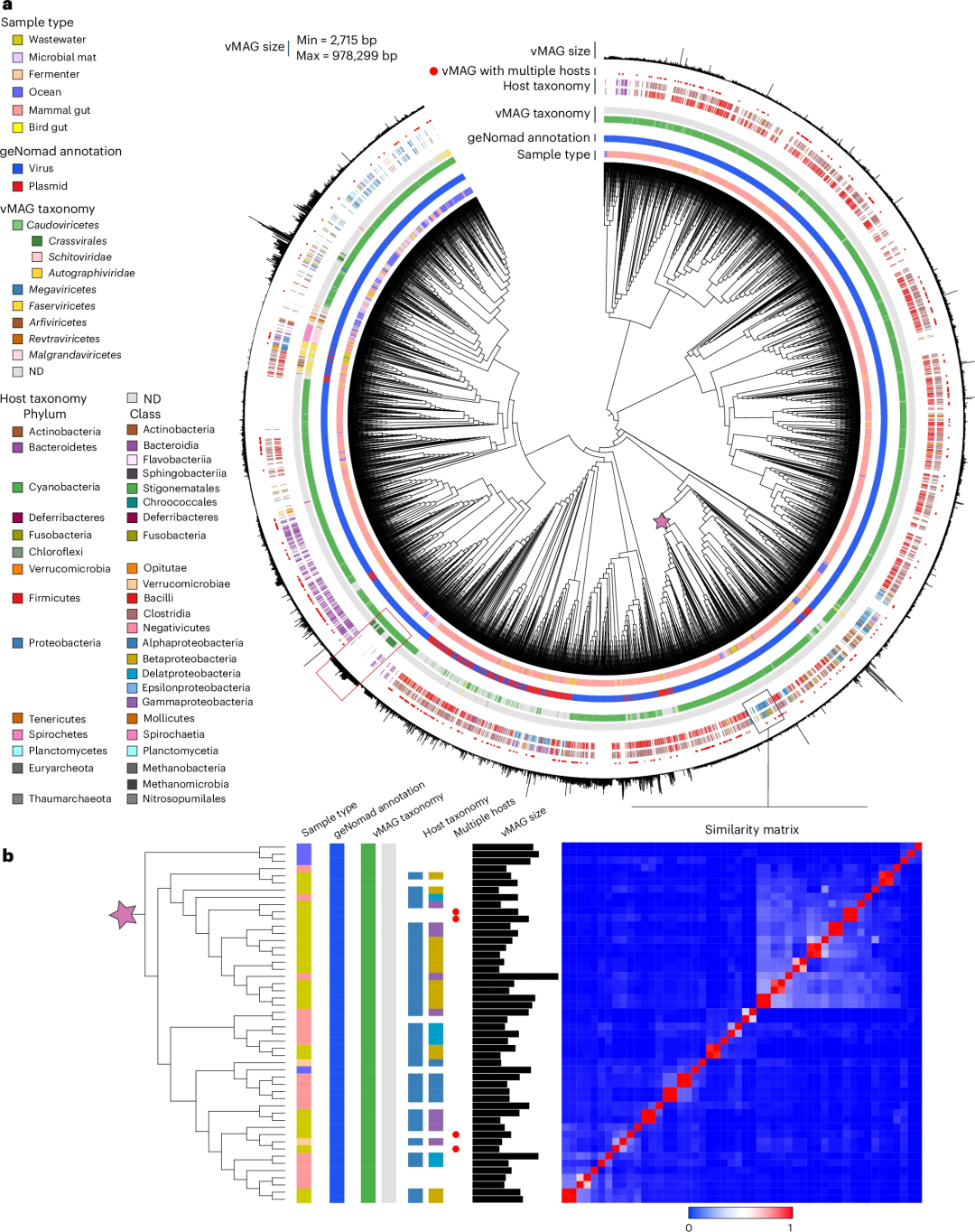
多宿主交互模式是一个常见特征
MetaTOR
研究开发了基于宏基因组三维组织的重组装流程,可简化宏基因组 metaHiC 数据集的处理和分箱。可以应用于已经测序的物种,也可以直接用于从头组装、支架和表征未知基因组的三维组织。
# 安装,conda是安装最新版本的官方推荐方式。
conda create -n metator bioconda::metator
# 激活环境
conda activate metator
# 使用
metator {network|partition|validation|pipeline} [parameters]
metator 命令采用以下形式metator action –param1 arg1 –param2 arg2 #etc.
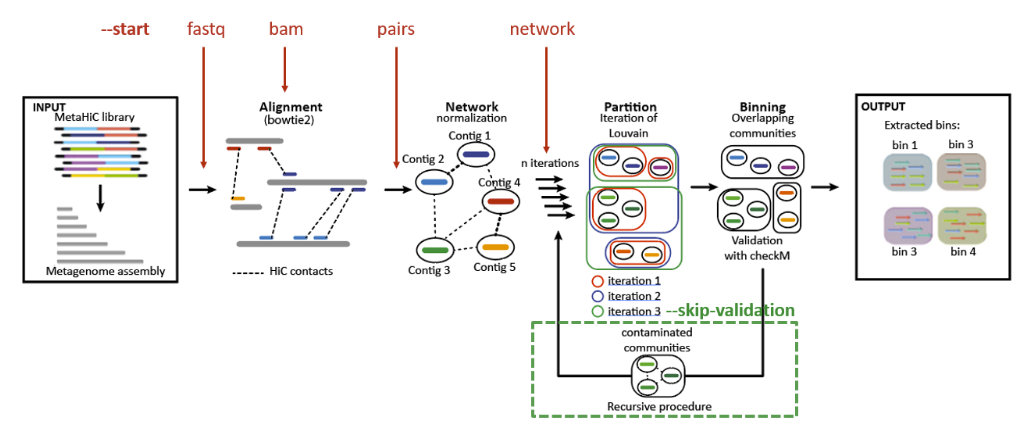
metator 管道中有三个作/步骤,必须以以下顺序运行:
-
• network:从 fastq 读取或 bam 文件生成 metaHiC 重叠群网络并将其归一化。 -
• partition:多次执行鲁汶或莱顿群落检测算法,根据重叠群之间的metaHiC信号对重叠群进行分箱。 -
• validation:使用 CheckM 验证垃圾箱,然后执行递归去污步骤以去除污染。
还有许多其他可选的杂项作:
-
• pipeline:根据给定的参数,按顺序运行上述所有三个作或仅运行其中的一些作。这可能需要一段时间。 -
• contactmap:从metator的最后一个输出的一个bin生成接触图。 -
• version:显示当前版本号。 -
• help:显示帮助消息。
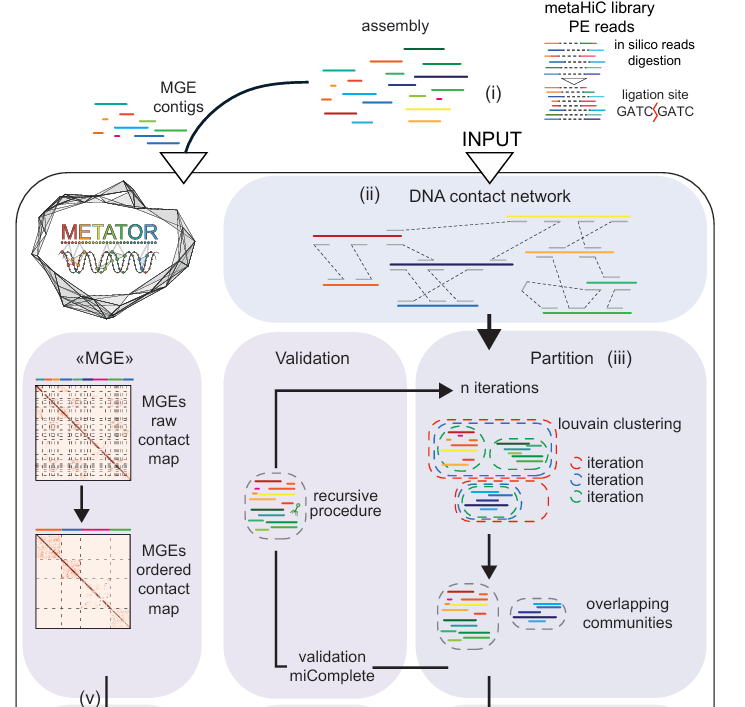
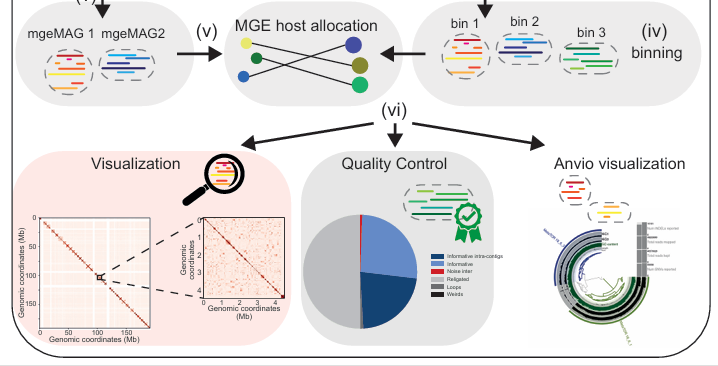
详细原理如下图:
参考
-
• 1.https://github.com/koszullab/metaTOR -
• 2.Bignaud, A., Conti, D.E., Thierry, A. et al. Phages with a broad host range are common across ecosystems. Nat Microbiol (2025). https://doi.org/10.1038/s41564-025-02108-2 -
• 3.https://research.pasteur.fr/fr/software/meta3c-metahic/
本篇文章来源于微信公众号:微因