试用了两个软件用于测试CNV检测,虽然没有取得什么结果,记录和分享一下。
ichorCNA笔记
这个软件可以检测切除的肿瘤组织,识别其中的肿瘤细胞含量,也可以用来检测纯肿瘤组织。可以有参考,也可以不用,官方提供了参考,可以自建。
1、 软件安装
软件官网:https://github.com/broadinstitute/ichorCNA
library(devtools)
install_github("broadinstitute/ichorCNA")
2、软件使用
# 1、准备数据,分块10K
hmmcopy_utils/bin/readCounter --window 10000 --quality 20
--chromosome "chr1,chr2,chr3,chr4,chr5,chr6,chr7,chr8,chr9,chr10,chr11,chr12,chr13,chr14,chr15,chr16,chr17,chr18,chr19,chr20,chr21,chr22,chrX,chrY"
/media/vd1/fastq/WGS/WGS-CNV/fastq/75bp_trim/sLK-1054_75_sorted.bam > 1054_75.wig
# 2、应用
Rscript ./runichroCNA.R --id 1054_75
--WIG 1054_75.wig --ploidy "c(2)" --normal "c(0.5,0.6,0.7,0.8,0.9)" --maxCN 5
--gcWig /media/vd1/zjd/Miniconda3/lib/R/library/ichorCNA/extdata/gc_hg19_10kb.wig
--mapWig /media/vd1/zjd/Miniconda3/lib/R/library/ichorCNA/extdata/map_hg19_10kb.wig
--centromere /media/vd1/zjd/Miniconda3/lib/R/library/ichorCNA/extdata/GRCh37.p13_centromere_UCSC-gapTable.txt
--normalPanel /media/vd1/zjd/Miniconda3/lib/R/library/ichorCNA/extdata/HD_ULP_PoN_1Mb_median_normAutosome_mapScoreFiltered_median.rds
--includeHOMD False --chrs "c(1:22)" --chrTrain "c(1:22)"
--estimateNormal True --estimatePloidy True --estimateScPrevalence True --txnE 0.9999 --txnStrength 10000
--scStates "c()" --outDir ./
参数说明:
1) –id 样本名
2)–WIG 前一步准备的样本文件
3) –gcWig GC含量的文件,illumina测序对GC含量有偏好,需要去除影响
4) –mapWig 这是参考基因组的10K window图谱
5) –centromere 每个染色体都有的着丝粒,去除
6) –normalPanel 阴性参考,可选,有的话去噪效果好,可自己用阴性构建
7) –includeHOMD 特别大Bin时如1M需要
8) –estimateScPrevalence FALSE –scStates “c()” 针对体细胸样本时需要,亚克隆 9) –chrs “c(1:22)” –chrTrain “c(1:22)” 只训练和分析常染色体
10) –estimateNormal True 评估正常的,这个参数默认,可以不加
11) –estimatePloidy True 评估肿瘤细胞倍性,也是默认,可不加
12) –estimateScPrevalence True 亚克隆情况,体细胞需要,可设置False
3、结果
以3个阴性样本为对照,生成Normal 参考,去噪,衡量两个阳性样本,发现只有一个缺失较大片段(20K+,s1054)的结果可以看出,只是图不太一样,并不能看到明确缺失。
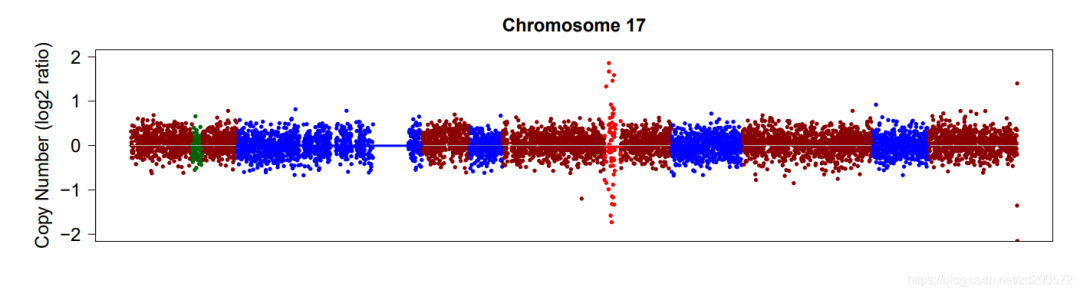
4、参考Panel构建
可以使用阴性对照建立自己的基线参考,不限样本,越多越好,去噪有一定效果,这里采用10K窗口进行构建:
#panel
Rscript createPanelOfNormals.R
--filelist ./noraml.txt
--gcWig /media/vd1/zjd/Miniconda3/lib/R/library/ichorCNA/extdata/gc_hg19_10kb.wig
--mapWig /media/vd1/zjd/Miniconda3/lib/R/library/ichorCNA/extdata/map_hg19_10kb.wig
--centromere /media/vd1/zjd/Miniconda3/lib/R/library/ichorCNA/extdata/GRCh37.p13_centromere_UCSC-gapTable.txt
--outfile normal_sy
二、 QNDASeq笔记
1、软件安装
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("QDNAseq")
BiocManager::install("QDNAseq.hg19")
2、软件使用
#增加内存限制,防止不足,不设置会报错
options(future.globals.maxSize=5000000000)
library(QDNAseq)
bins <- getBinAnnotations(binSize=5)#获取每一个bin的基因注释,联网下载
bins
readCounts <- binReadCounts(bins,bamfile='sLK-1054_75_sorted.bam')
readCounts
pdf("readCounts.pdf")
plot(readCounts, logTransform=FALSE, ylim=c(-50, 200))
highlightFilters(readCounts, logTransform=FALSE,residual=TRUE, blacklist=TRUE)
readCountsFiltered <- applyFilters(readCounts,residual=TRUE, blacklist=TRUE,chromosomes=c("1","2","3","4","5","6","7","8","9","10","11","12","13","14","15","16","18","19","20","21","22","X", "Y"))
dev.off()
pdf("mapping.pdf")
isobarPlot(readCountsFiltered)
readCountsFiltered <- estimateCorrection(readCountsFiltered, ncpus=8)
dev.off()
pdf("sd.pdf")
noisePlot(readCountsFiltered)
copyNumbers <- correctBins(readCountsFiltered)
copyNumbers
copyNumbersNormalized <- normalizeBins(copyNumbers)
copyNumbersSmooth <- smoothOutlierBins(copyNumbersNormalized)
dev.off()
pdf("cnr.pdf")
plot(copyNumbersSmooth)
exportBins(copyNumbersSmooth, file="sample.txt")
exportBins(copyNumbersSmooth, file="sample.igv", format="igv")
exportBins(copyNumbersSmooth, file="sample.bed", format="bed")
dev.off()
######1.3Downstream analyses#########
copyNumbersSegmented <- segmentBins(copyNumbersSmooth, transformFun="sqrt")
copyNumbersSegmented <- normalizeSegmentedBins(copyNumbersSegmented)
pdf("cns.pdf")
plot(copyNumbersSegmented)
copyNumbersCalled <- callBins(copyNumbersSegmented)
dev.off()
pdf("cnc.pdf")
plot(copyNumbersCalled@featureData@data)
exportBins(copyNumbersCalled, format="vcf")
exportBins(copyNumbersCalled, format="seg")
dev.off()
总结,没有可靠结果,软件暂时认为不可用于10Kb以下的CNV检测,特别是1x这种测序深度。
本篇文章来源于微信公众号: 微因