最近又搜索了下基因型填充的相关内容,之前一直没找到一个超级详细的一步步做的教程,于是就使用impute + pipeline 这个关键词搜索了百度、必应和谷歌,发现流程还不少,虽然有些可用性存疑。这里强调的是最后一个,broad研究所出品,应该必属精品,有没有兴趣一试呀!了解好原理,使用这些大神的脚本,站在巨人的肩膀上,解决自己的能力短板,也不错,轮子不必总重复造。 流程发表于2019年末,还是比较新的。
流程发表于2019年末,还是比较新的。
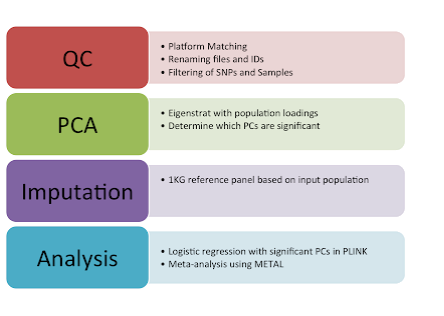 从上图可以看出,它有四个功能呢,填充只是它的一小部分。
从上图可以看出,它有四个功能呢,填充只是它的一小部分。
分享一下,查找到的资料:1、Genotype Imputation — SNP & Variation Suite v8.9.0 Manual (goldenhelix.com) 个人DTC基因检测的填充
2、Genome-wide imputation pipeline — genipe 1.5.0 documentation (pgxcentre.github.io) 没有参考的制作
3、Getting Started – Michigan Imputation Server 之前用的docker
4、SHAPEIT (ox.ac.uk)
5、IMPUTE2 (ox.ac.uk)
6、使用IMPUTE2进行基因型填充 (360doc.com)
7、汉族基因组数据库 (hanchinesegenomes.org) 国内的万人参考,可在线填充
8、File format reference – PLINK 2.0 (cog-genomics.org)
9、GitHub – transbioZI/Gimpute: An efficient genetic data imputation pipeline 没有参考的制作 2018
Gimpute: an efficient genetic data imputation pipeline | Bioinformatics | Oxford Academic (oup.com)
10、Genetics GwasImputePipeline.pdf – Array Suite Wiki (arrayserver.com) 2016 没有参考的制作
11、GitHub – lifebit-ai/snp-imputation-nf: Nextflow script to create the imputation 没有参考的制作 2018
12、赵栋/molgenis-imputation (gitee.com) 没有参考的制作,不再维护 ImputationPipeline_old – bbmri
13、GitHub – VertebrateResequencing/pbwt: Implementation of Positional Burrows-Wheeler Transform for genetic data Sanger
14、GitHub – Orion1618/Odyssey: Odyssey: A semi-automated pipeline for the prepping, cleaning, phasing, imputing, analyzing, and visualizing genomic data 2019没有参考的制作
15、bioinformatics/impute2-pipeline.pl at master · johnlees/bioinformatics · GitHub 2016
16、MitoImpute: A Snakemake pipeline for imputation of mitochondrial genetic variants (biorxiv.org) MtDNA
17、90392999 (biorxiv.org) 工具比较shapeit-impute2完胜
18、https://github.com/ryhui/imputation-pipeline 2020
19、Ricopili 这个官网要科学的上网呀 https://sites.google.com/a/broadinstitute.org/RICOPILI/home
有没有使用经验的一起交流下呀?
阅读原文,是带链接的笔记。
扫描二维码
获取更多精彩
公众号

本篇文章来源于微信公众号: 微因