之前有个软件SNP2HLA可以把snp芯片的结果转化成HLA分型的,软件数据库里面还有中国人的数据集,相对准确,可以分到每个基因座两位,准确度因基因座而不同,还有个网站,也可以实现将23andme等的结果提取出HLA型。下面我测试一下:
1、网站
网站的方法,简单易行,虽然可能准确度有点问题,先试试。http://hla.nicokist.com/
之前找到过一次,现在找不到了,github是个东西,终于在上面找到了,ps他最近被微软收购了,不知未来如何啊。话说这个网页是采用HIBAG这个R包实现的,看名字应该是个中国人或者华人写的,点赞一个!
话说这个芯片的数据有部分是sgi开头的,估计是公司自己加上去的,不清楚是什么含义的情况下,先用一个超级简单的python脚本去掉,脚本如下:
fout = open('ywc-drop-sgi.txt', 'w')
with open('ywc.txt') as f:
for line in f:
if line.startswith('sgi'):
continue
else:
fout.write(line)
fout.close()
好吧,还是不行,去掉没有分出结果的‘–’的位点,还是报错。很遗憾地表示,不管怎样修改数据格式,就是说不能读取23andMe数据(Error: Could not read 23andMe data file.),只好放弃了。看它的示例数据只有A、B、C三个基因座的结果,结果准确性偏低的。
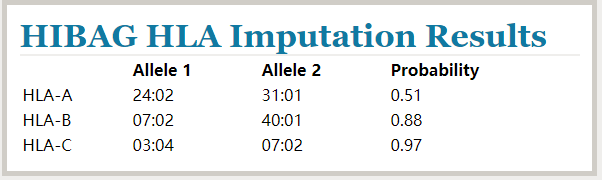
今天逛wegene论坛又发现一个可以将wegene的数据转换成23的,试试行不行,地址地这:http://joshua.galaxy.42dna.com/wgto23/
竟然成功了,结果类似上面的,虽然只有01, 0.59和0.85的准确率。
2、SNP2HLA
这个软件安装比较简单的,下载下来直接用。