顾名思义,MetaSUB (http://metasub.org/)项目就是研究地铁和城市宏基因组,已扩展到许多不同领域的多个项目。不仅在地铁上进行了采样,还在纪念碑、下水道、海滩、空气和医疗环境中以及全球一百多个城市进行了采样。但是项目网站证书已经过期,感觉已经疏于维护啦,不过看采样网站什么的还在更新,论文看到有2021年发表的Cell。采样网站:https://portal.geoseeq.com/sample-groups/20ffce9b-f3f8-4ac8-b0d5-e2a85772b08a,看到还有一些中国城市的样本,北京,台北等。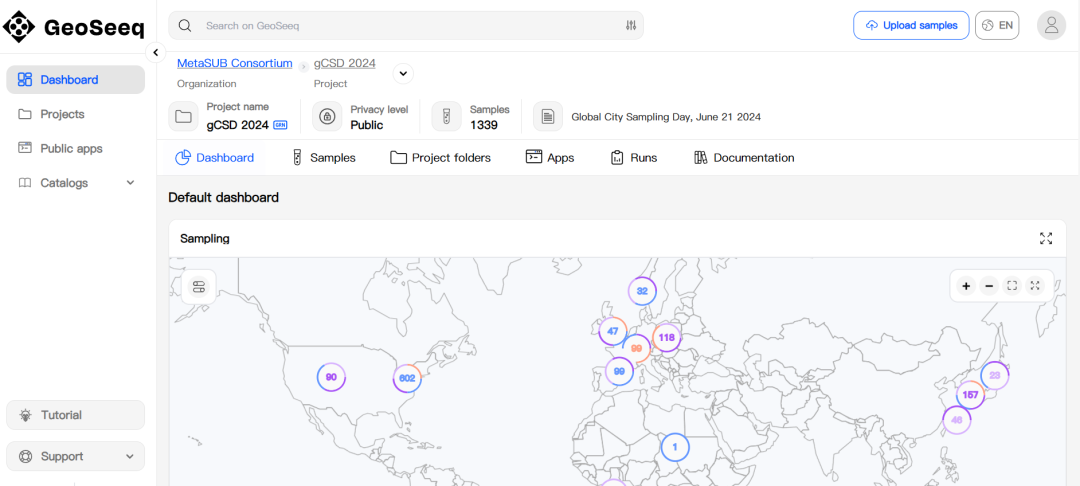
MetaSUB 的主要目标
-
创建地理空间宏基因组和法医遗传图谱
-
识别和跟踪城市建筑环境中的抗菌素耐药性标志物 (AMR)
-
鉴定用于药物发现的新型生物合成基因簇 (BCG,MetaBGC)
生信分析方面
除了我们比较熟悉的人类微生物组计划开发的MetaPhlAn,HUMAnN, PanPhlan和QIIME等,还有所说在临床用的SURPI等,也有自己的分析流程https://github.com/MetaSUB/CAP2。也是有原始数据可供下载的,需要登陆才可以下载。
MetaSUB Core Analysis Pipeline 是一个用于宏基因组分析的 python 包。它是复杂管道上的一致界面,可以轻松构建更复杂的分析。它是应该在每个宏基因组上运行的核心工具集。它具有高度可重复性和可扩展性。它旨在消除分析微生物组的苦差事,同时使复杂的分析变得容易。
学习下SUPRI
SUPRI (基于序列的超快速病原体鉴定,http://chiulab.ucsf.edu/surpi, https://github.com/chiulab/surpi)主要用来对临床样品的 shotgun metagenomics 测序数据中寻找并鉴定病原的工具,也就是通常说的mNGS项目啦,我们知道项目时效性是相当强的。它有2种模式:Fast mode 和 Comprehensive mode。前者可以快速对测序的 reads 进行 Mapping,找到细菌或病毒 reads。后者分析更为详细,除了细菌病毒外还比对了真菌,寄生虫等其他物种数据库,并且进行de novo assembly,对contig也进行 Mapping,虽然耗时更长,但可以获得覆盖度,taxonomic 分类等信息。 从官网来看就是个华人学者的实验室,看衣服上的标志还有NASA的字样。该软件的临床版本 SURPI 目前被用作加州大学旧金山分校正在开发的传染病临床宏基因组下一代测序检测的核心分析管道,了解了下应该是商业化收费的,也可以理解啦。
会议
还有MetaSUB会议,最近一次是在日本东京,看到还有华东师范大学的中国学者参会。
其他
自 2016 年以来,每年 6 月 21 日都会采集样本。扩展的项目有:METACoV:RNA/COVID-19 环境采样,METAMED:全球医院抽样,METACATS:家猫冠状病毒和微生物组,奥林匹克运动会:奥运会抽样,StuckOnU (“粘”在手机上的 DNA,以查看微生物组以预测手机所有者最后吃的东西是什么,预测他们的祖先),METWOSEW:下水道和废水采样,METABEA:海滩和海岸线采样。
好的,今天就分享到这啦!
本篇文章来源于微信公众号:微因