百万退伍军人计划 (MVP) 这个我们相对比较熟悉了,当然在中美竞争的大背景下,想要像获得UKB等原始数据进行分形的可能性是基本没有了,但是还可以看看公开的数据,看有没有什么有价值的内容。这两天从Nature文章中发现一个网页,感觉不错,分享一下!
百万退伍军人计划 (MVP)
先来复习一下这个计划,退伍军人事务部 (VA) 的百万退伍军人计划 (MVP) 是美国最大的退伍军人数据基因组生物库,拥有世界上所有基因研究计划中最多样化的队列之一。来自参与的退伍军人的数据正在帮助研究人员了解基因、生活方式和军事经历如何影响健康和保健。由于我们与数百名研究人员的合作以及他们的合作,MVP 数据已经导致了有关焦虑、创伤后应激障碍 (PTSD)、心脏病、肾脏疾病、癌症等疾病的新发现。
CIPHER
美国退伍军人事务部 (VA)他能源部合作 开发,是一个在线知识共享平台,旨在优化电子健康记录 (EHR) 数据以用于研究和临床作,称为集中式交互式表型组学资源 (CIPHER)。是世界上最大、最多样化的全基因组 x 全表型组关联研究 (gwPheWAS)。
方法和结果
在 635,969 名美国退伍军人中展示了与千人 基因组计划中的非洲人 (123,328)、美国混血儿 (61,111)、东亚人 (7,076) 和欧洲人 (457,720) 超级种群基因相似的人群分层现象范围的 GWAS,然后进行多人群荟萃分析。报告了 1,274 个性状中的 38,270 个独立变异,精细映射将 6,318 个信号从 613 个性状缩小到单变异分辨率。其中,在欧洲人口之外发现了 2,069 个关联,这证明了扩大遗传研究多样性的重要性。
结果探索
PheWeb
PheWeb 由密歇根大学开发,旨在探索大型遗传数据集的关联结果,包含百万退伍军人计划 (MVP) 全基因组 PheWAS 的结果。分析了 2,068 个特征,其中包含几千名亚裔美国人。有两个分析结果,协调血统和种族/民族 [HARE],它结合使用自我报告和遗传信息,以及 遗传推断血统 [GIA]。
不过开放程度和中国的CKB有些类似,基本只限于网页上的位点的展示,详细信息需要申请。GRCh38,只显示大约 5000 个Top位点,请访问 dbgap 获取完整结果
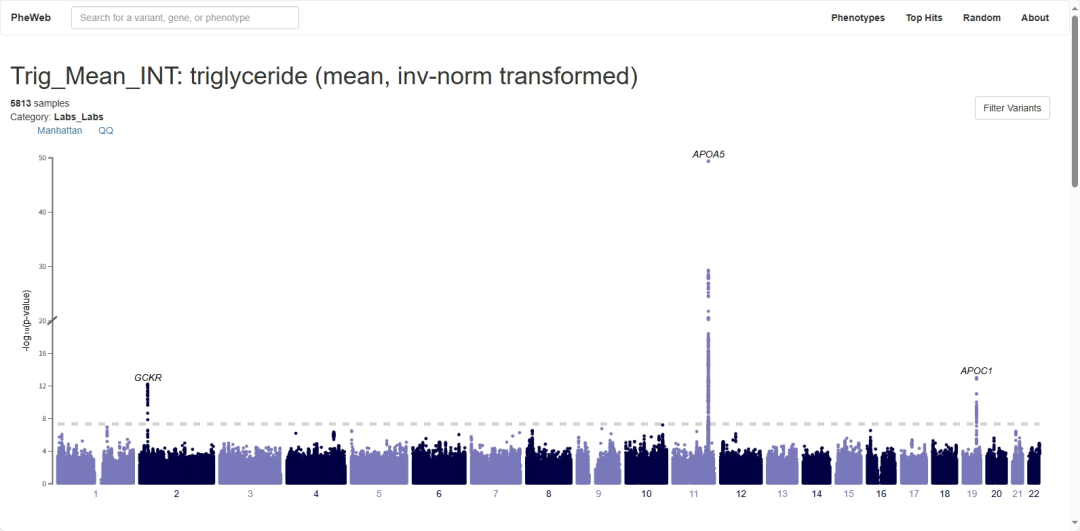
全基因组全表组关联研究 (gwPheWAS)
一个比较明显的R语言Shiny app,按人群提供 SNP 与表型之间关联的可视化;此外,还允许比较跨种群的结果,以浏览遗传位点和表型之间潜在异质关系的结果。可以按 SNP 或特征进行搜索,选择感兴趣的结果并比较跨祖先的结果。
当前版本包括单一性状 GWAS 发现、单一 SNP PheWAS 发现的可视化,以及这些发现在人群中的比较,突出了少数族裔人群和欧洲人群之间存在显着差异的 SNP 性状对。
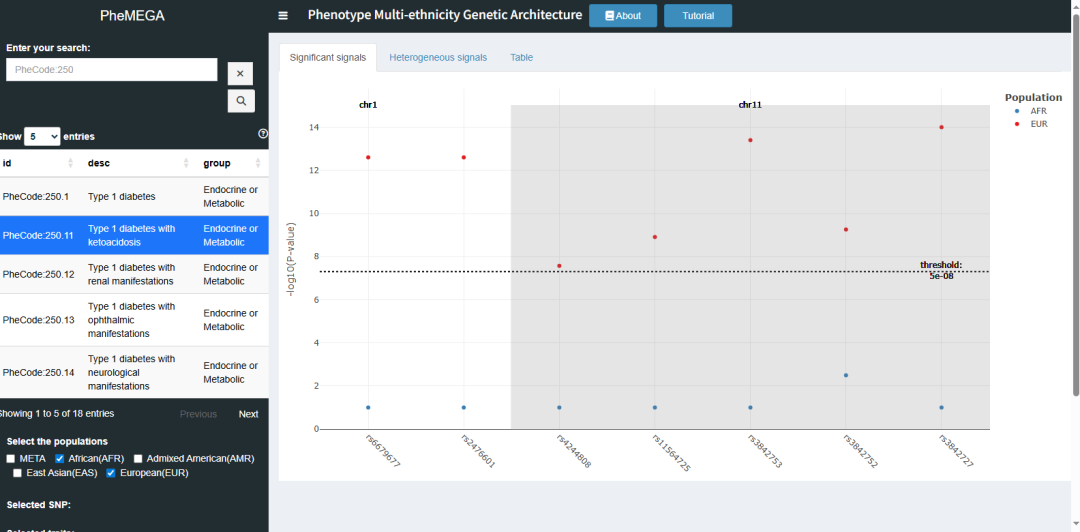
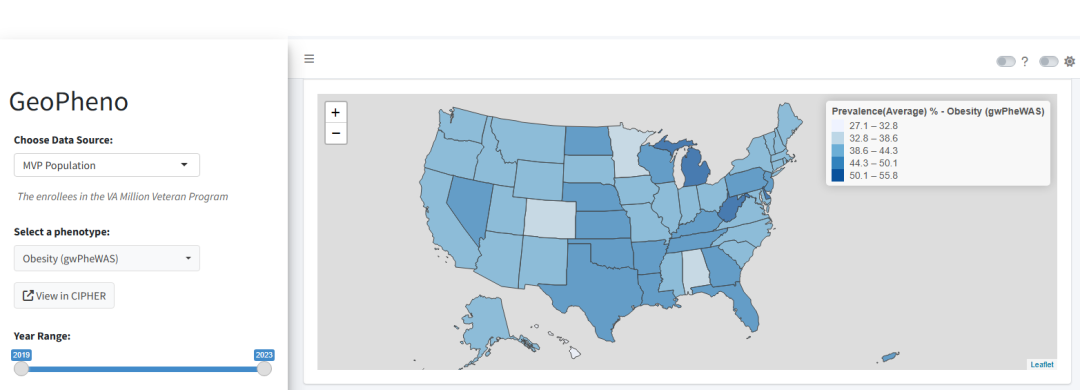
GeoPheno
地理空间可视化工具显示随时间变化的条件患病率。显示一段时间内使用基于电子健康记录 (EHR) 的表型定义的健康状况的地理空间流行率。
药物发现功效靶点
可视化和总结孟德尔随机化生成的结果。Druggable Genome Shiny 应用程序是一种药物靶点发现工具,旨在可视化和总结孟德尔随机化生成的结果,合成多源、多模态汇总级数据,包括来自全基因组关联研究和 eQTL/pQTL 研究的数据。显示各种表型和全基因组变异之间的关联分数,使研究人员能够有效地获取信息、交叉检查多种表型并识别与其中几种表型相关的基因。
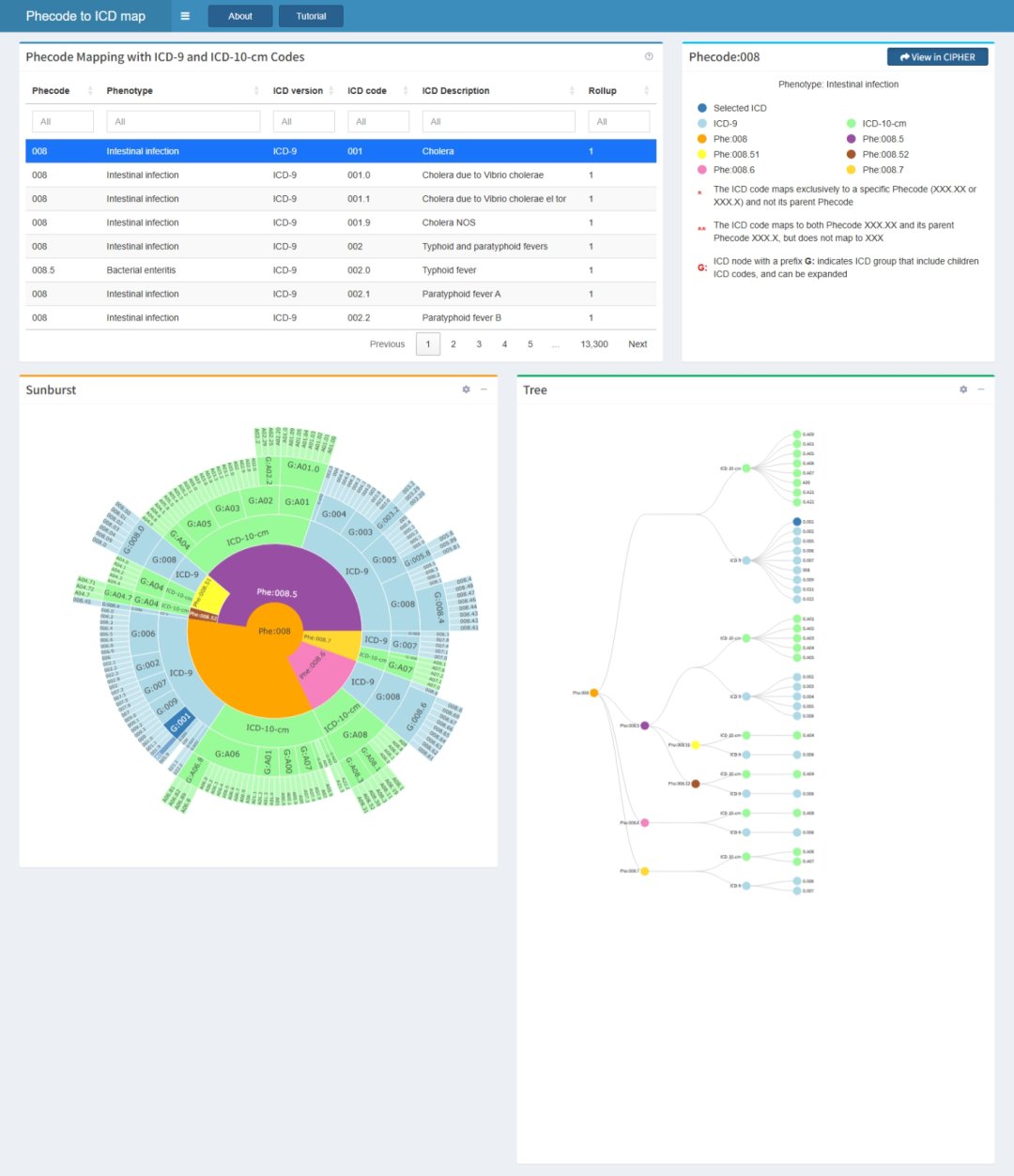
Phecode to ICD map
基于电子健康记录 (EHR) 的研究在研究中具有多种优势:它们具有成本效益,允许进行大规模纵向分析,并有可能分析数百种人类疾病、药物反应和许多可观察到的临床特征。可视化从 Phecode 到国际疾病分类 ICD-9 和 ICD-10 代码的映射。
还有个可视化的交互式知识图谱,推断疾病、治疗、程序和实验室测量之间的相关性,但是不公开,需要账号才能进入。

本篇文章来源于微信公众号:微因