🌍 背景痛点:传统方法为何低效?
1. 双倍成本:原核生物(细菌、古菌)用16S rRNA,真核生物用18S rRNA,需分开测序,耗时烧钱。
2. 偏差难控:通用引物易导致扩增偏差,长序列测序误差大,真核生物基因拷贝数差异干扰结果。
3. 数据库割裂:现有数据库(如SILVA、PR2)仅专注某一类,叶绿体等细胞器序列常被误判为“未知”。
🛠️ 解决方案:CABO-16S数据库
其有三大亮点:
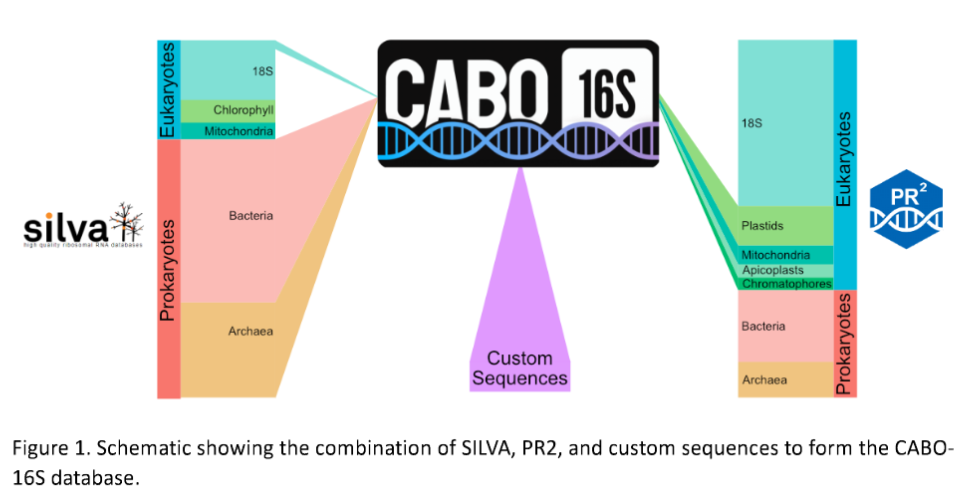
1. 三合一整合:
-
细菌 & 古菌:来自SILVA数据库(38万+序列)。
-
真核细胞器(叶绿体、线粒体等):来自PR2数据库(8540条序列)。
-
自定义序列:支持用户添加特定环境或类群的专属数据(如深海甲烷渗漏区的SEEP-SRB1菌)。
-
2. 智能分类算法:
-
基于IDTAXA算法,高精度匹配序列。
-
全长度16S rRNA训练,避免截断导致的模糊分类。
3. 灵活扩展性:
-
未来可无缝更新SILVA和PR2新版本。
-
用户可针对性添加小众环境微生物数据,提升分类分辨率。
📊 实验结果:分类性能全面碾压!
1. 光合成真核生物识别率飙升:
-
在海洋、湖泊等富光合环境,CABO-16S的ASV(扩增子序列变体)分类数比SILVA提升10%。
-
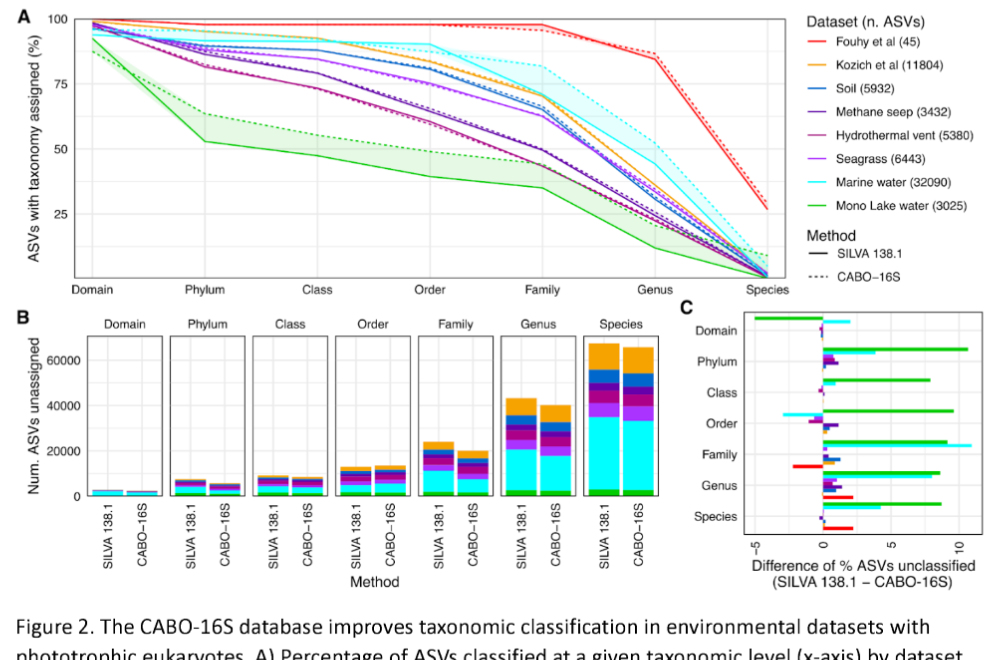
-
案例:Mono湖样本中,50%的读长被精准归类为真核叶绿体。
2. 细分复杂类群:
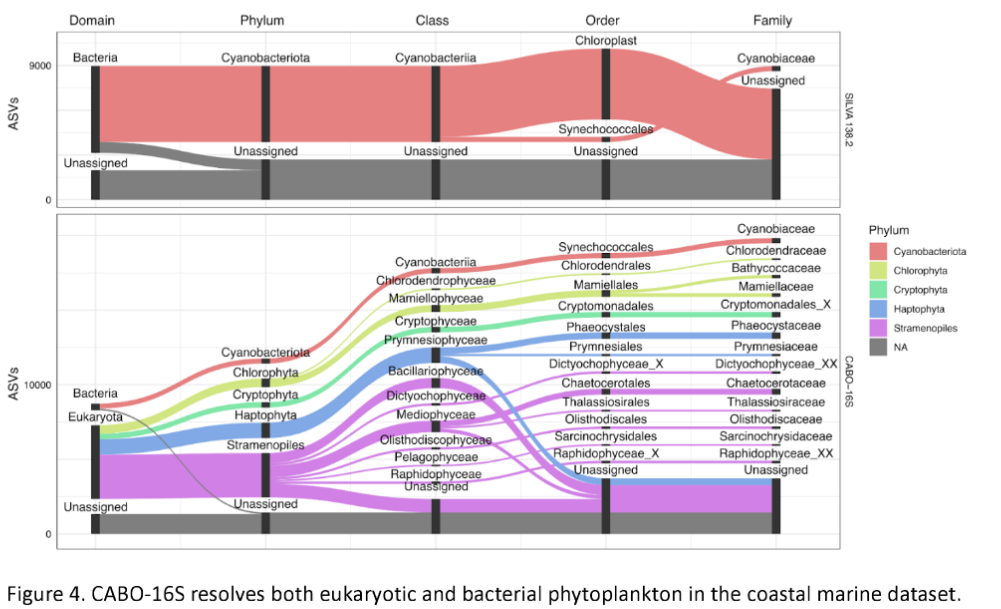
-
SEEP-SRB1菌群:传统数据库仅能识别到“属”,CABO-16S通过添加自定义序列,区分出共生型与非共生型亚群,助力深海甲烷氧化机制研究。
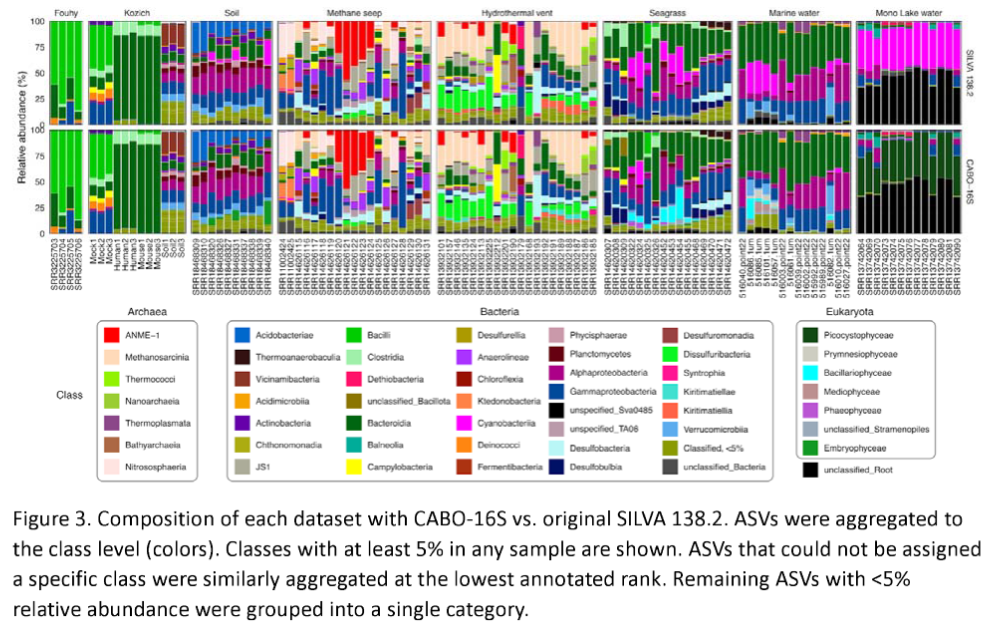
3. 兼容性与稳定性:
-
非光合样本中,细菌/古菌分类准确率与SILVA持平,验证数据库通用性。
-
🚀 应用场景:谁需要CABO-16S?
-
环境微生物组研究:海洋、湖泊、土壤等复杂样本的微生物群落解析。
-
极端环境探索:深海热泉、甲烷渗漏区等特殊生态系统的功能菌群鉴定。
-
生态与气候研究:光合微生物群落动态监测,助力碳循环模型构建。
🌟 未来展望:更智能、更全面
-
扩充线粒体16S序列(当前PR2中96.7%为后生动物来源)。
-
优化多源数据兼容性,解决分类学与系统发育学的命名冲突。
📚 免费获取:
-
GitHub代码:https://github.com/emelissa3/CABO-16S
-
自定义序列库:Figshare数据
结语:CABO-16S像一把“万能钥匙”,解锁了微生物世界的隐藏地图。无论是科研大牛还是新手,都能用它高效探索微观生命的奥秘!
本篇文章来源于微信公众号:微因