最近做PRS评分,需要用到连锁不平衡信息,来进行位点筛选,找了好几个工具用于计算连锁不平衡,也发现了个好用的网页工具来进行查询,在这里和大家分享一下!
地址在这,阅读原文也是这个,后面的操作都在这个网页进行的。由于这个网站用到了谷歌的一些组件,可能需要科学上网才能访问。什么,你不会,我有个凑活着用的方式,要不要试试,回复”setup”,给你个小工具。
LDlink
首页
先来欣赏下首页,美国NIH的癌症研究所的工具,还是比较权威的,数据来自千人基因组计划,可以分人种进行信息查询。可以看到有好几个工具可用,我一般用的是SNPclip,这个工具可以对同一条染色体的n个snp进行连锁不平衡的筛选。
可以看到它还是在更新的,最近一次更新在今年9月份。还可以用R包等api进行批量查询。

各个模块
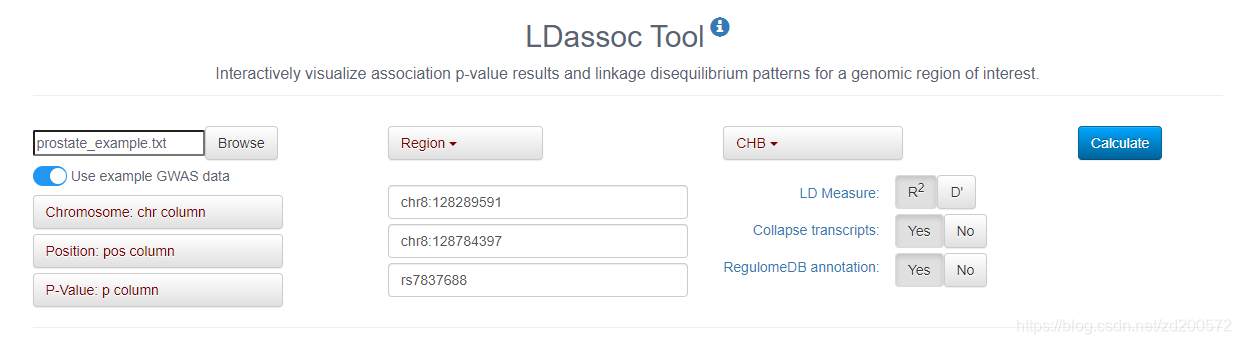
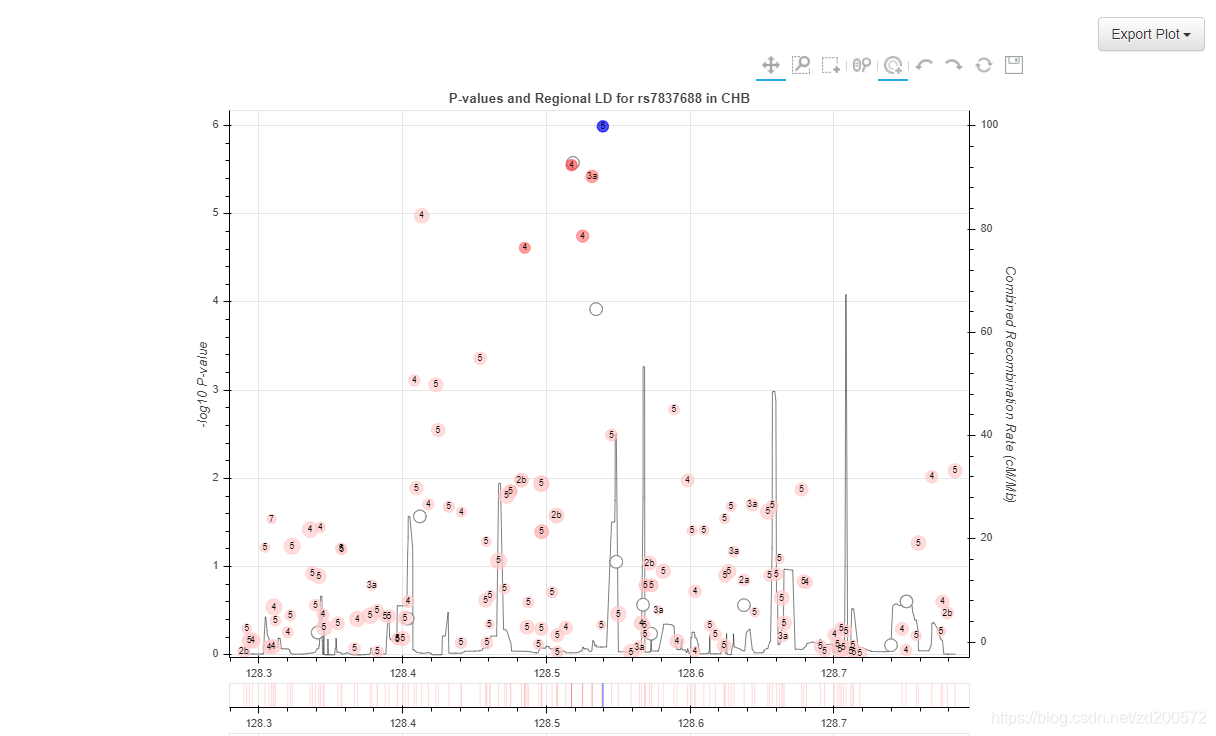
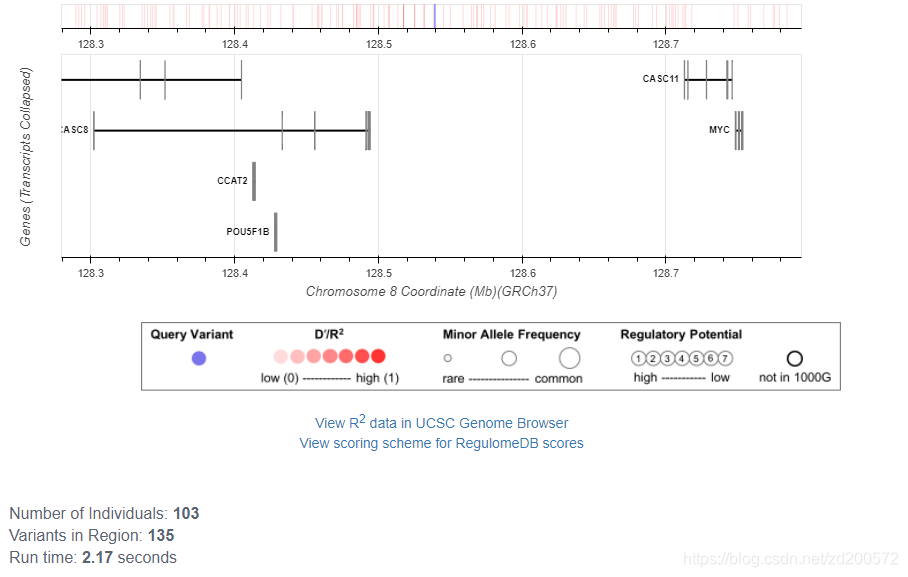
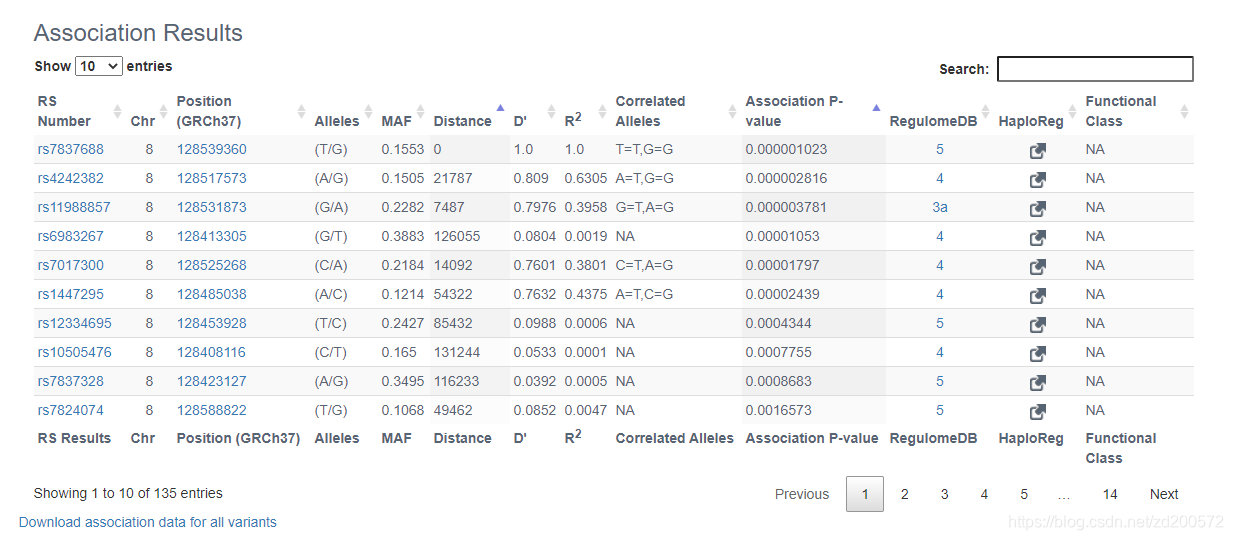
LDassoc Tool
交互地可视化感兴趣基因组区域的关联p值结果和连锁不平衡模式。
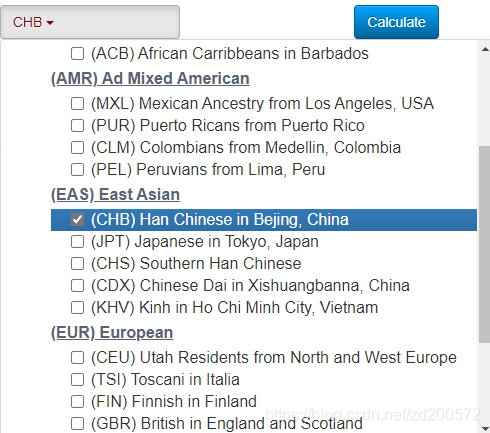
选择人种信息,点击Calculate, 就可以获得你所需的结果了。
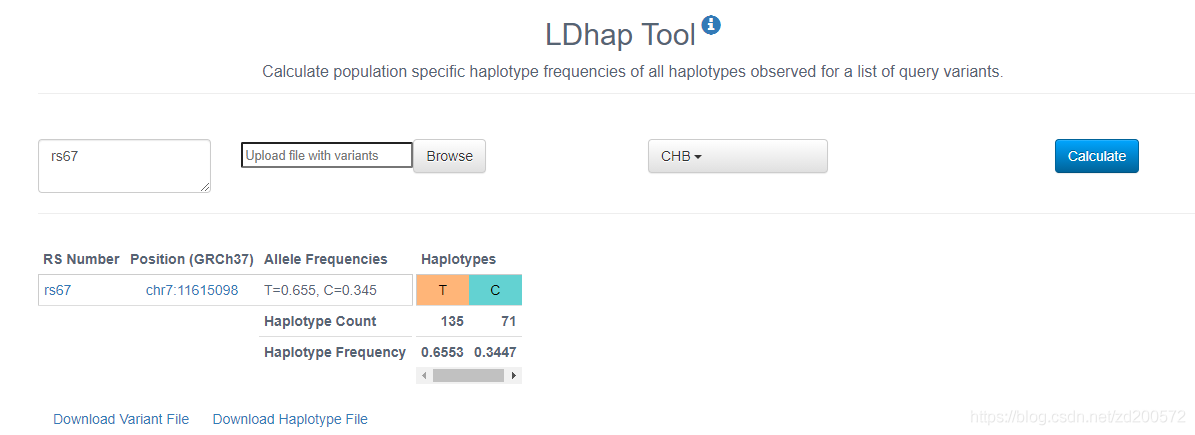
LDhap Tool
查询SNP的所有单倍型的群体特定单倍型频率。
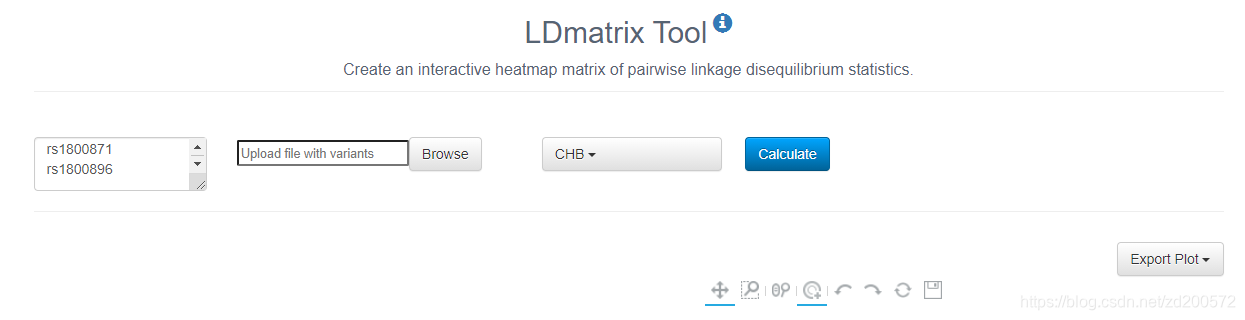
LDmatrix Tool
创建成对不平衡统计的交互式热图矩阵。


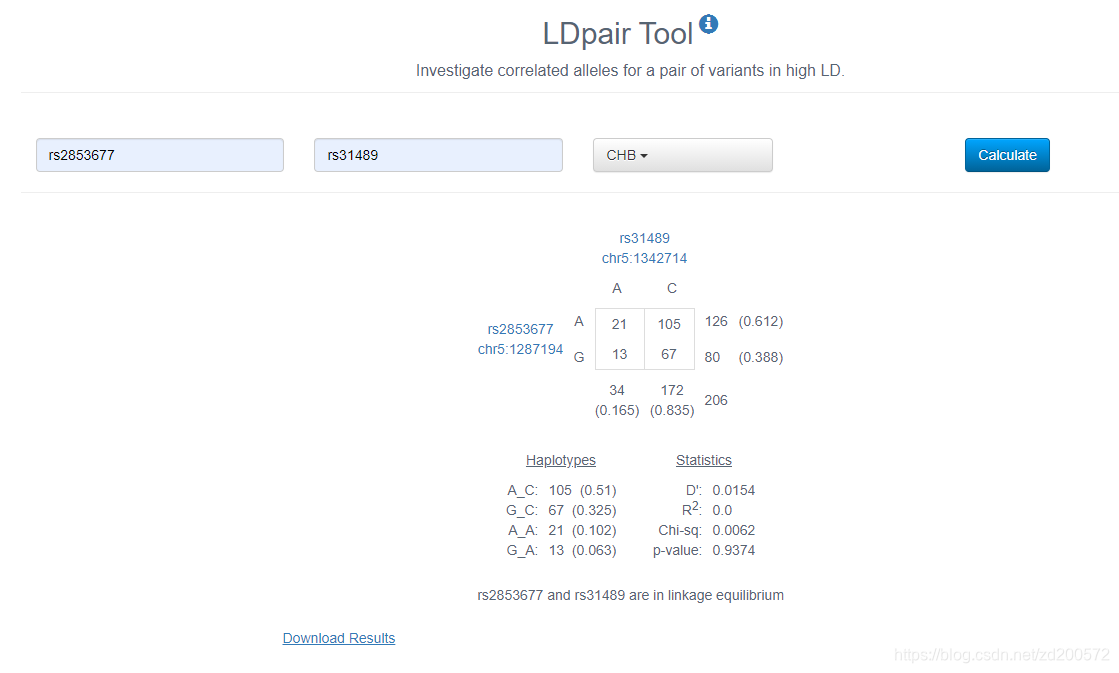
LDpair Tool
调查一对高LDSNP的相关等位基因。
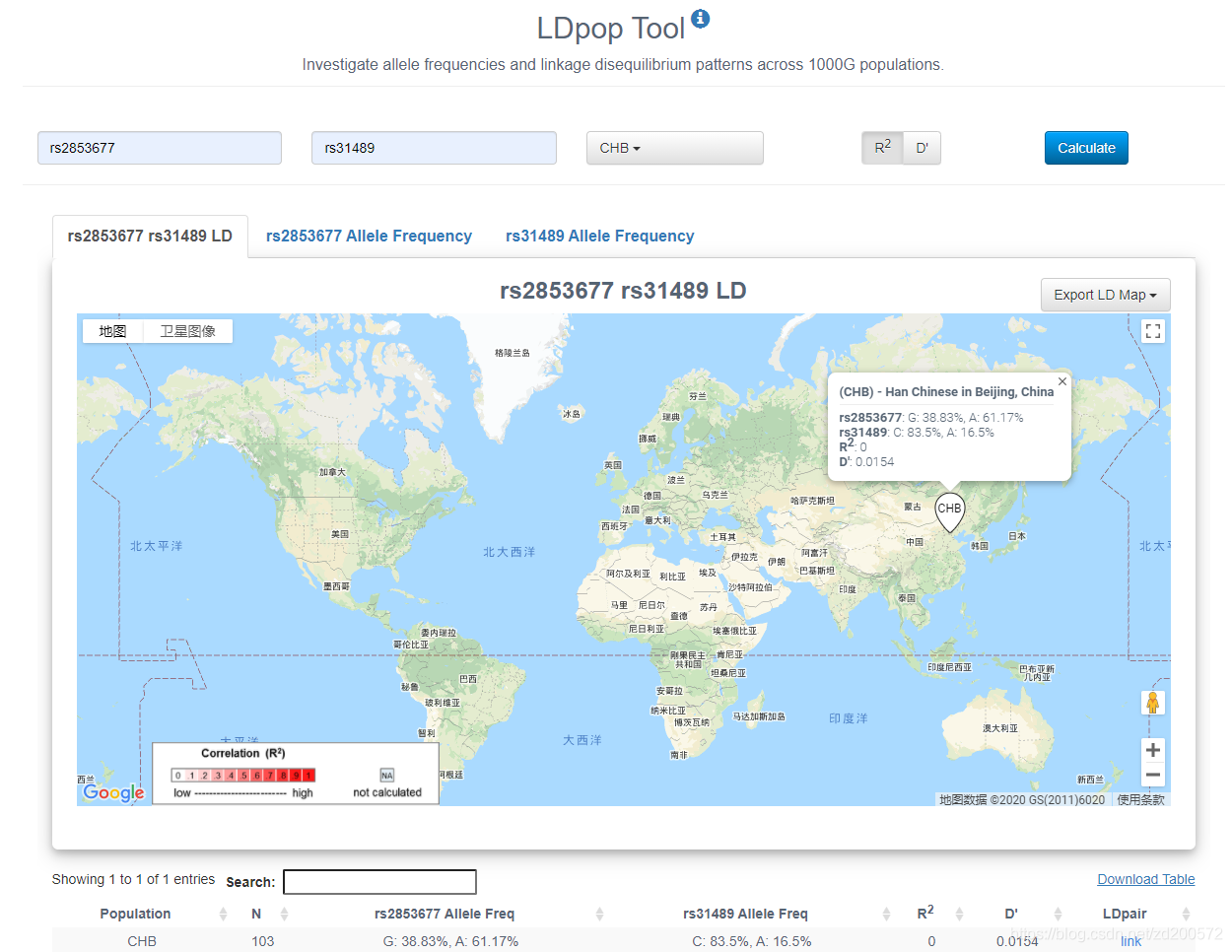
LDpop Tool
调查1000G人群中的等位基因频率和连锁不平衡模式。
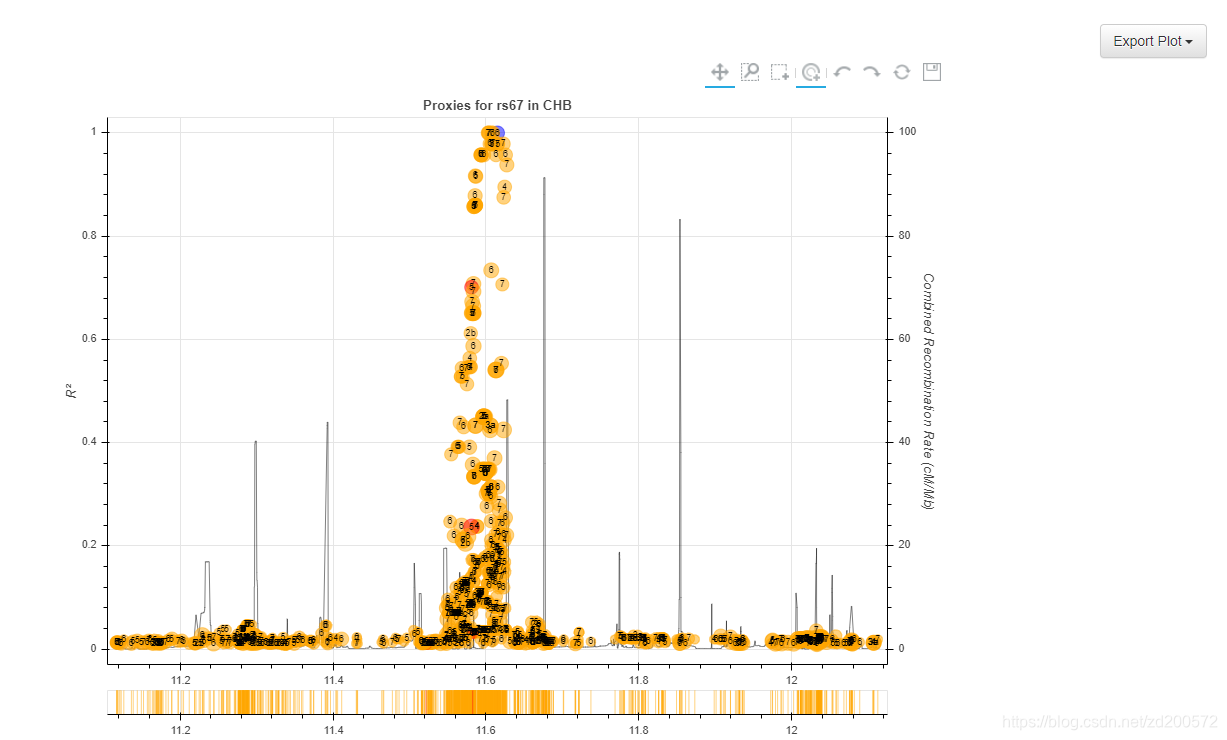
LDproxy Tool
交互式浏览查询SNP的代理SNP和假定功能。

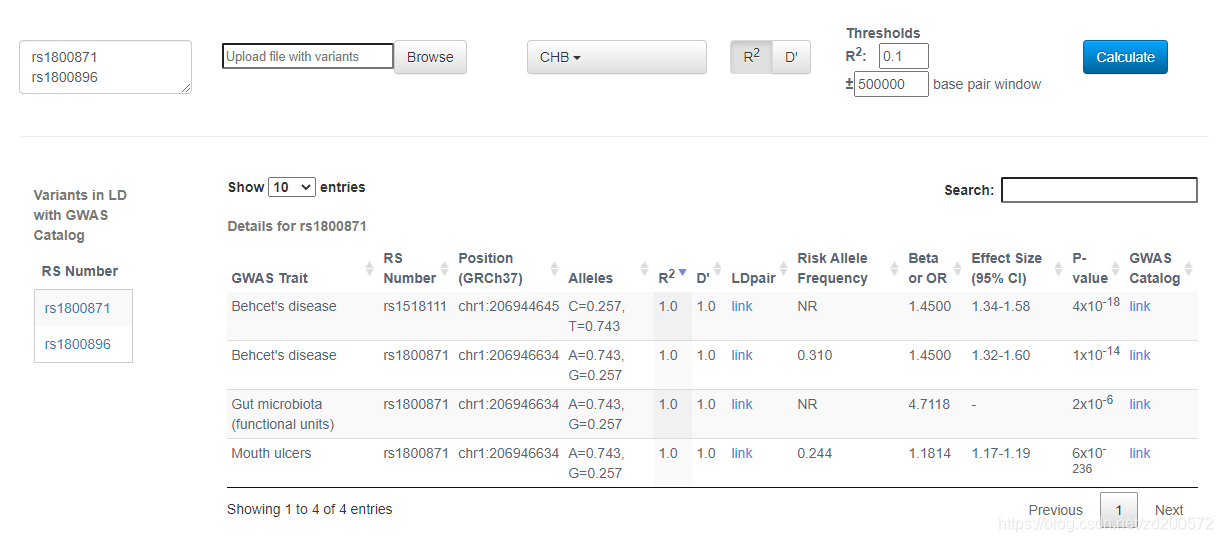
LDtrait Tool
顾名思义,是查询有没有相关疾病关联的研究。
SNPchip Tool
这个应该是用来选择SNP芯片的。
SNPclip Tool
我学得这个是筛选LD最常用的了。

厉害